 Dieser Artikel ist Teil des ScienceBlogs Blog-Schreibwettbewerb 2018. Informationen zum Ablauf gibt es hier. Leserinnen und Leser können die Artikel bewerten und bei der Abstimmung einen Preis gewinnen – Details dazu gibt es hier. Eine Übersicht über alle am Bewerb teilnehmenden Artikel gibt es hier. Informationen zu den Autoren der Wettbewerbsartikel finden sich in den jeweiligen Texten.
Dieser Artikel ist Teil des ScienceBlogs Blog-Schreibwettbewerb 2018. Informationen zum Ablauf gibt es hier. Leserinnen und Leser können die Artikel bewerten und bei der Abstimmung einen Preis gewinnen – Details dazu gibt es hier. Eine Übersicht über alle am Bewerb teilnehmenden Artikel gibt es hier. Informationen zu den Autoren der Wettbewerbsartikel finden sich in den jeweiligen Texten.
——————————————————————————————————————
Next-Generation-Sequencing – Wie man heutzutage DNA auslesen kann
Von Bernhard Scharinger
Ich studiere Biomedical Engineering an der Technischen Universität Graz und schreibe im Moment meine Bachelorarbeit über RNA-Sequencing. Dies ist mein zweiter Beitrag zum Schreibwettbewerb und ich freue mich auf die vielen anderen guten Beiträge.
Grundsätzlich besteht DNA (Desoxyribonucleic acid) aus einem Zucker (der Desoxyribose) und einem Phosphatrest, die das „Rückgrat“ der DNA bilden. Zusammen mit den vier verschiedenen Basen Adenin (A), Thymin (T), Guanin (G) und Cytosin (C) bilden sie das DNA-Molekül. Diese Basen sind unter anderem auch für den Informationsgehalt des Genoms, die Gen-Sequenz, verantwortlich. Als Frederick Sanger 1977 die Kettenabbruchmethode zur Bestimmung von Gen-Sequenzen entdeckte, revolutionierte seine Arbeit die Biologie und machte den Weg für die Forschung am Genom frei. Seitdem hat sich sehr viel getan in der Bioinformatik. Neue Methoden wurden entwickelt, um DNA-Sequenzen noch schneller und zuverlässiger zu bestimmen. Diese neuen Technologien, auch massive parallel sequencing- oder next generation sequencing – Methoden genannt, können große Mengen and Daten verarbeiten, und Genome in nur einigen Stunden vollständig sequenzieren. In diesem Beitrag möchte ich euch die Technik, die hinter der Sequenzierung steckt, näherbringen.
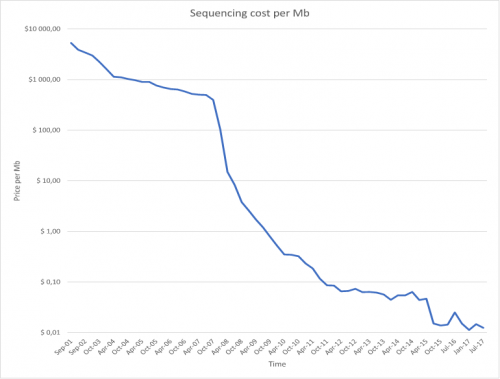
Die Kosten von DNA-Sequnzierung pro Megabase. Die Daten dazu stellt K. Wetterstrand vom Genome Sequencing Program (GSP) unter diesem Link zur Verfügung.
Die erste Technologie, die ich vorstellen möchte ist das so genannte Illumina/Solexa sequencing. Diese Technologie basiert auf der Methode der Sequenzierung mit Synthese und ist mit den verschiedenen Plattformen von Illumina einer der Marktführer im Bereich der Gen-Sequenzierung. Der erste Schritt dieser Methode ist die Vermehrung der DNA-Probe. Das besondere hier ist, dass Illumina im Gegensatz zu einigen Konkurrenten nicht die Methode der polymerase chain reaction (PCR) verwendet, sondern eine Technologie die bridge amplification genannt wird. Durch die hohe Anzahl an identischen DNA-Strängen, die man nach der amplification erhält, werden auch die Signale, die man im Sequenzierungs-Schritt erhält, verstärkt. Die Sequenzierung beginnt damit, dass zur DNA reversible Terminatoren hinzugefügt werden. Das sind Moleküle, die ein weiteres Sequenzieren verhindern. Diese Terminatoren sind mit einem fluoreszierenden Marker versehen, der entsprechend der jeweiligen Base eine andere Farbe hat. Nun beginnt eine Polymerase, ein Enzym zum Bau von DNA, damit, einen Terminator an das 3‘-Ende eines Primers zu hängen. Nach dem Anhängen eines Nukleosids wird der ganze Prozess gestoppt (terminiert – daher der Name) und alle anderen Nukleoside werden weggewaschen. Anschließend werden mit einem Laser die fluoreszierenden Marker erregt, und das emittierte Licht kann gemessen werden. So weiß man, welche der 4 verschiedenen Basen gerade angehängt wurde. Der ganze Ablauf wird so lange wiederholt, bis die gesamte zu sequenzierende Sequenz gelesen wurde. Hierzu gibt es ein sehr anschauliches Video auf Youtube in Englisch:
Eine neuere, sehr faszinierende Methode der Sequenzierung ist das Pacific BioSciences sequencing, oder auch PacBio-Sequencing. Anders als viele andere Technologien, benötigt PacBio keine amplification der DNA vor der Sequenzierung. Es handelt sich um eine single-molecule-real-time-Technologie (SMRT). Diese Methode zählt zu den Sequencing-Technologien der dritten Generation und ist eine sehr vielversprechende Entwicklung. Das Herzstück dieser Methode ist ein so genannter zero-mode waveguide (ZMW), ein ca. 70-100 Nanometer (etwa 1000-mal weniger als die Dicke eines Haars!) tiefes Loch, an dessen Boden eine einzelne DNA-Polymerase angebracht ist. Hier läuft die Sequenzierung folgendermaßen ab: Als aller erstes werden an die Enden der doppelsträngigen DNA Adaptoren angebracht, die die Form einer Haarnadel haben. Entwirrt man den DNA-Strang nun ergibt sich ein einzelner kreisförmiger DNA-Strang welcher SMRTbell genannt wird. Nun gibt man eine SMRTbell in einen einzelnen ZMW und die Polymerase bindet an einen der Adaptoren. Im nächsten Schritt werden die wieder fluoreszierend markierten Nukleotide hinzugefügt, diesmal jedoch ohne einen terminierenden Teil. Der Prozess kann nun gestartet werden und die Polymerase beginnt damit, mit den vorhandenen Basen den komplementären Strang zu bauen. Es wird wieder Licht verwendet, um die fluoreszierenden Markierungen zum leuchten zu bringen, aber dadurch, dass der Durchmesser der ZMWs wesentlich kleiner ist, als die Wellenlänge des Lichts, wird nur der Boden des ZMWs beleuchtet. Das führt dazu, dass immer nur die Base, die gerade verbaut wird, zum Leuchten angeregt wird. So erhält man einen „Film“ von Lichtimpulsen mit unterschiedlichem Spektrum, wobei jedes dieser Spektren einer einzelnen Base zugeordnet werden kann. Sieht man sich den Film nun an, weis man ganz genau, wie die Sequenz unserer DNA-Probe lautet. Auch zu dieser Technologie gibt es Videomaterial, das Pacific Biosciences auf Youtube geladen hat:
Die dritte Technologie, die ich euch vorstellen möchte, ist das Oxford Nanopore Sequencing. Ähnlich wie die Methode von PacBio, kann mit Oxford Nanopore Sequencing ein einzelnes Molekül in Echtzeit sequenziert werden. Außerdem erfordert diese Form der Sequenzierung auch keinen Amplification-Schritt am Anfang. Das Besondere an dieser Technik ist eine Polymer-Membran mit sehr kleinen Poren (Nanopore), die gerade so groß sind, dass ein einzelner DNA-Strang durch sie hindurch passt. An die Membran wird eine Spannung angelegt, und die geladenen Moleküle in den durch die Membran getrennten Bereichen beginnen, durch die Membran zu fließen. Da DNA negativ geladen ist, passiert auch sie die Pore. Fließt ein Molekül durch die Nanopore, so verursacht das einen Spannungsabfall der Membranspannung, der je nach Molekül unterschiedlich ist. Mit speziellen Algorithmen kann nun der Spannungsverlauf, der entsteht, wenn ein DNA-Strang die Pore passiert, dekodiert werden und man erhält die Information über die Sequenz des Moleküls. An die Pore wird zusätzlich noch ein Motor-Protein angebracht, das dazu dient, die DNA zu entwinden und die Geschwindigkeit, mit der sie durch die Pore fließt zu steuern. Oxford Nanopore hat hierzu ein gutes Video online gestellt:
Jede der hier vorgestellten Technologien hat ihre Vor- und Nachteile. Ein wichtiger Punkt sind die Kosten für die Sequenzierung. So kostet zum Beispiel das Instrument PacBio RS im Moment fast 700.000$, sodass sich nur große oder gut geförderte Labore diese Technologie leisten können. Zum Vergleich: Das Startpaket für den Oxford Nanopore MinION kostet 1.000$ und ist somit sehr gut für kleine Labore oder einzelne Personen geeignet. Allerdings ist beim MinION die Qualität der Ergebnisse noch wesentlich schlechter als zum Beispiel beim etablierten Illumina MiSeq oder PacBio RS. Allgemein können wir erwarten, dass in Zukunft die Kosten für die Sequenzierung immer weiter sinken werden und die Qualität der Sequenzierung immer weiter steigen wird, sodass vielleicht bald jeder in seiner Garage mal schnell das Genom seiner Katze bestimmen kann. Ähnlich zu den riesigen Firmen, die aus interessierten Studenten in einer Garage entstanden sind, könnten in Zukunft vielleicht große Durchbrüche in der Genomik in einfachen Heim-Laboren in Kellern oder Garagen passieren. Zusammenfassend lässt sich sagen, dass das Next-Generation-Sequencing vielleicht eine der wichtigsten Entwicklungen in der Biologie in den letzten Jahren war. Die erste Next-Generation-Sequencing-Plattform kam 2005 auf den Markt, man könnte fast sagen, diese Technologien stecken noch in ihren Kinderschuhen. Mit der ständigen Weiterentwicklung vorhandener Technologien, und der Entwicklung neuer Technologien können wir Großes in der Zukunft erwarten.
Quellen:
D. Clark und N. Pazdernik. Molecular Biology Elsevier Inc., Waltham, MA, Second edition, 2013
B. Erguner et al. Performance comparison of Next generation sequencing platforms. Proceedings of the Annual International Conference of the IEEE Engineering in Medicine and Biology Society, EMBS, volume 2015-November, pages 6453-6456, 2015
J. Shendure und H. Ji, Next-generation DNA sequencing. Nature Biotechnology , 26(10):1135-1145, 2008
M. Xiong et al. Next-Generation sequencing. Journal of Biomedicine and Biotechnology , 2010:1-2, 2010











Kommentare (6)