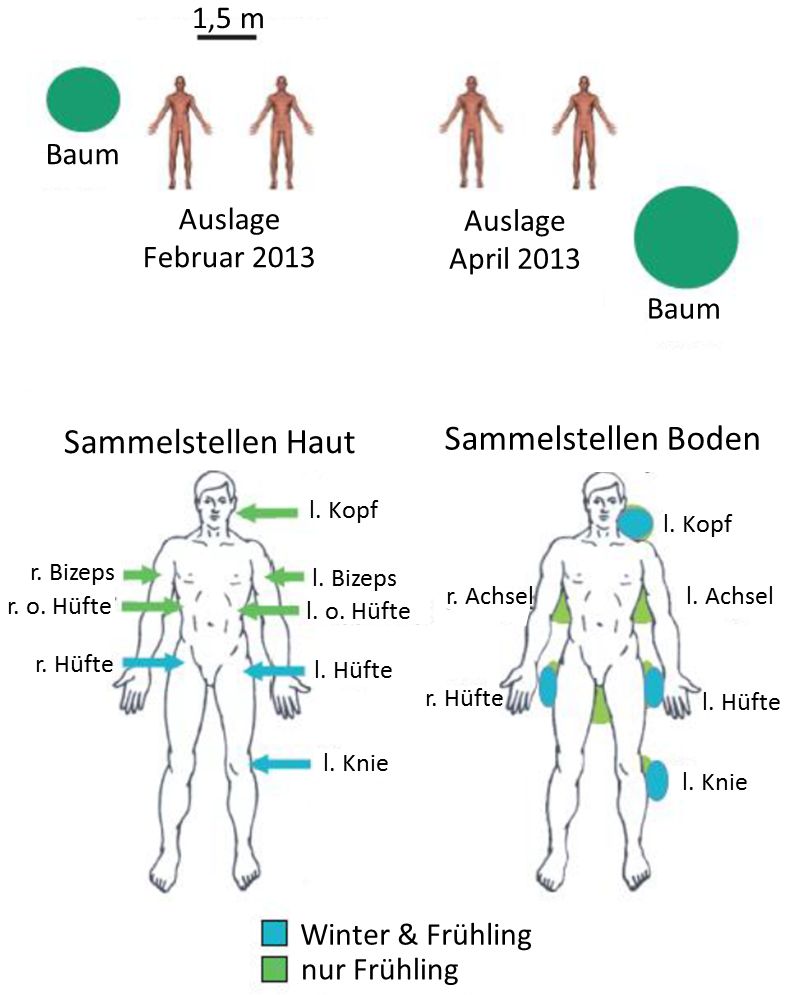
Die Körper gehörten Menschen, die sie zu Lebzeiten der Wissenschaft gespendet hatten und die Abbildung zeigt, wie die Leichname ausgelegt wurden und wann und von welchen Stellen der Leichname und des Bodens, auf dem sie lagen, Proben genommen worden waren. Die Leichname waren allen möglichen Umwelteinflüssen ausgesetzt und waren auch frei zugänglich für tierische Aasfresser (Geier waren diesmal, glaube ich, aber nicht dabei ;-). Über eine Dauer von 143 und 82 Tagen wurden regelmäßig (einen Monat lang täglich oder alle zwei Tage, danach in größeren Zeitabständen) Proben von Haut und Boden (hier bezeichnet als „Graberde“) genommen.
Aus den Proben wurde die DNA extrahiert und mittels NGS-Analyse von 16S- und 18S-rRNA sowie interner transkribierter spacer-Regionen die Zusammensetzungen der Metagenome der Gemeinschaften von Archaea, Bakterien, Einzellern und Pilzen dokumentiert und über die Zeitpunkte der Probennahmen hinweg verglichen.
![Lorarithmisch skalierte Heatmap (Häufigkeitsdarstellung) operationaler taxonomischer Einheiten (OTUs) auf der Haut menschlicher Leichen. Jede Zeile entspricht einem Taxon. Man erkennt, wie sich mit der Zeit die Häufigkeit der verschiedenen Taxa verändert und wie ähnlich das entstehende Muster bei verschiedenen Leichen ist. aus [1]](https://scienceblogs.de/bloodnacid/files/2016/05/B.jpg)
Lorarithmisch skalierte Heatmap (Häufigkeitsdarstellung) operationaler taxonomischer Einheiten (OTUs) auf der Haut menschlicher Leichen. Jede Zeile entspricht einem Taxon. Man erkennt, wie sich mit der Zeit die Häufigkeit der verschiedenen Taxa verändert und wie ähnlich das entstehende Muster bei verschiedenen Leichen ist. aus [1]
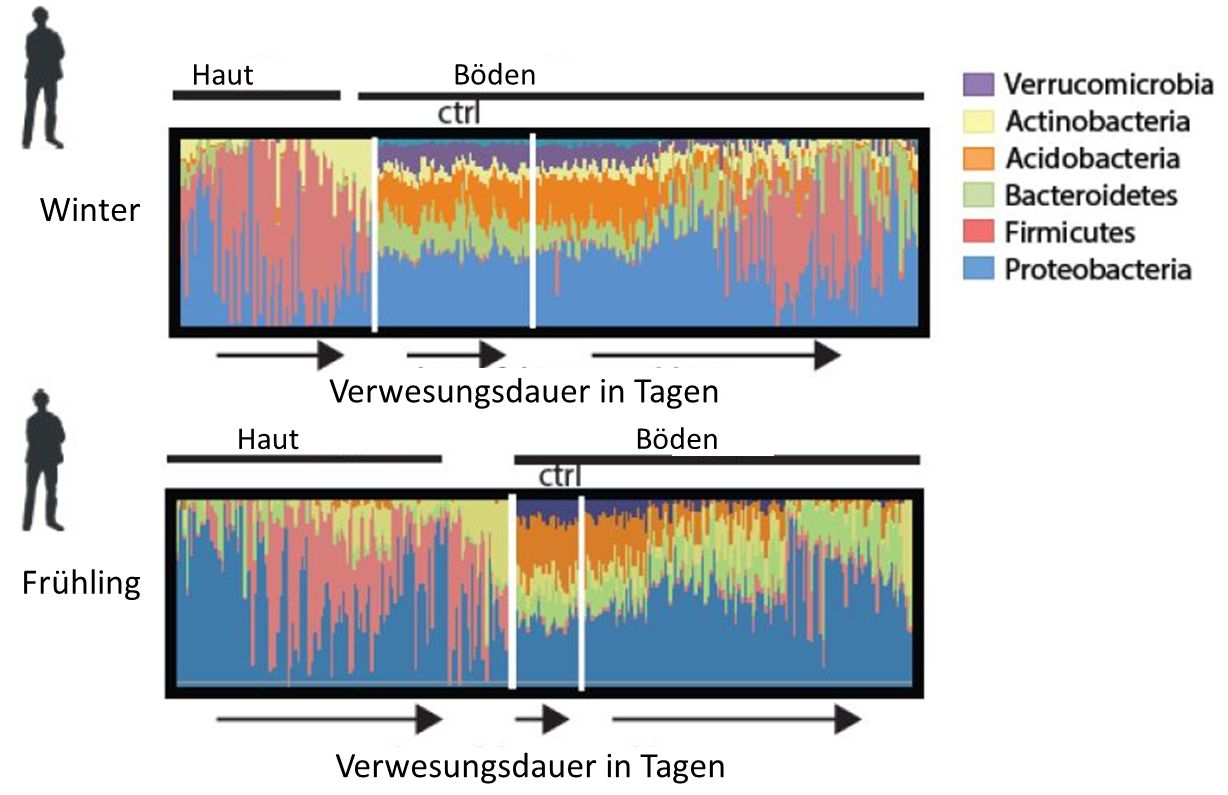
Relative Häufigkeit von Bakterienstämmen, charakterisiert anhand von 16S-rRNA-Daten auf menschlichen Leichen und assoziierter Graberde im Winter und Frühling. Die Proben sind gruppiert nach Probentyp und angeordnet nach Verwesungsdauer (Tage seit Deponierung). Jeder Balken repräsentiert die relative Häufigkeit der Bakterienstämme (farbig codiert) in einer einzelnen Probe. Stämme mit einer Häufigkeit < 0,1% sind hier nicht angezeigt. aus [1]
Dabei zeigte sich, daß die Abfolgen und Änderungen der Zusammensetzungen der gesammelten Mikrobiome so regelhaft und übrigens unabhängig von der Jahreszeit verliefen, daß sie sich mathematisch modellieren ließen und so eine Berechnung des PMI gestatteten. Anhand ähnlicher Versuche an Mauskadavern konnten die Gruppe zudem nachweisen, daß auch die Art des Bodens, auf der der Leichnam liegt, keinen Einfluß auf diesen Effekt hat.
Um das PMI zu berechnen, erstellten die Forscher ein Random-Forest-Regressions-Modell (ich habe das auch schon mal erklärt), das es ermöglichte, das PMI und damit den Todeszeitpunkt mit einer Genauigkeit von wenigen Tagen zu berechnen:
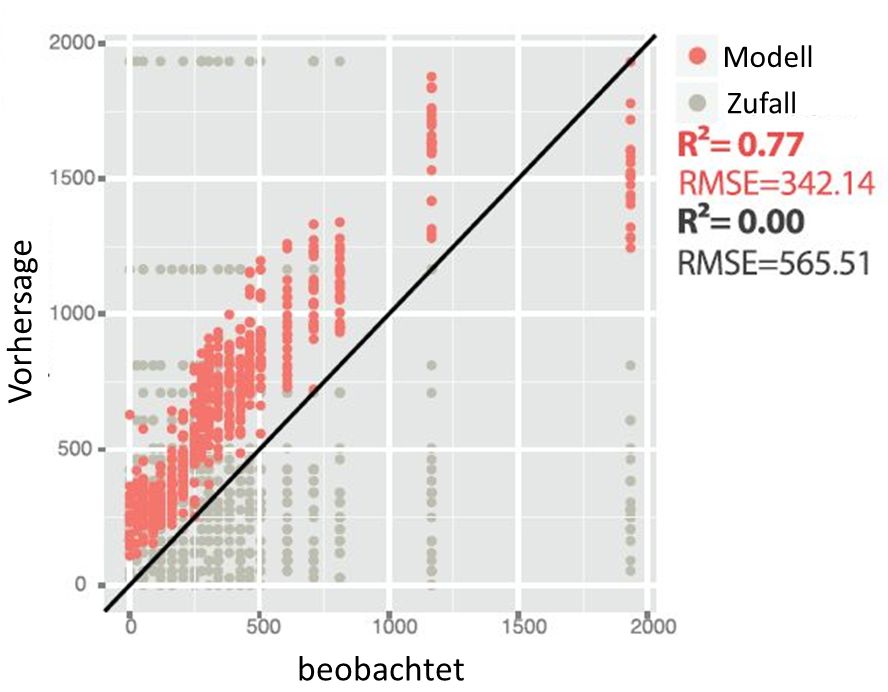
16S-rRNA-Daten-basiertes Random-Forest-Modell (RFM), das mit den Daten der Winter-Leichen trainiert wurde, um die PMI der Frühlings-Leichen zu berechnen. Jeder Punkt repräsentiert eine Probe, die nach einem bestimmten PMI genommen wurde, wobei die RFM-berechneten Werte in rot und zufällig vorhergesagte Werte in grau erscheinen, RMSE: root mean square error; aus [1]
Die Forscher probierten auch aus, welches Probennahme-Intervall die beste Vorhersagegenauigkeit ergab, indem sie die 16S-rRNA-Daten der Frühlings-Leichen in entsprechende Entnahmeintervalle unterteilten. Sie fanden, daß tatsächlich die tägliche Probennahme die genauesten Vorhersagen ermöglichte:
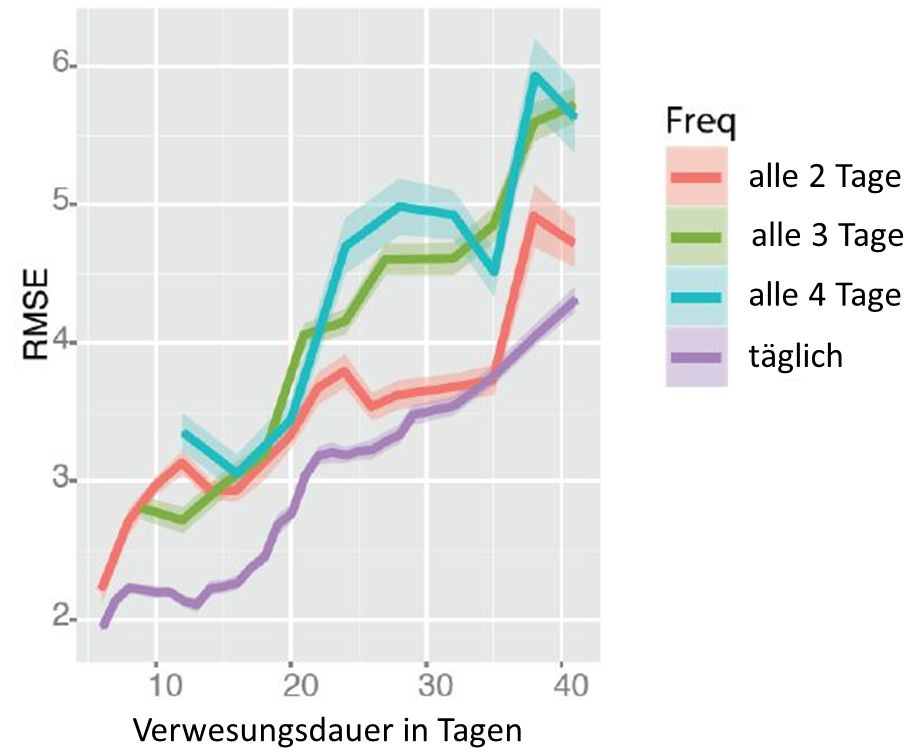
RMSE (engl. root mean square error), ist eine Maßzahl zur Beurteilung der Prognosegüte. Sie gibt an, wie gut eine Funktionskurve an vorliegende Daten angepasst ist, beziehungsweise wie stark eine Prognose im Durchschnitt von den tatsächlichen Beobachtungswerten abweicht; aus [1]
Über die Herkunft der mikrobiellen Destruenten läßt sich aufgrund weiterer Daten sagen, daß ca. 40% aller Taxa schon zu Beginn der Experimente in sehr geringen Mengen im Boden vorhanden waren, womit der Boden die Hauptquelle für mikrobielle Destruenten ist. Die Forscher fingen aber sogar 79 leichenbesiedelnde Fliegen und untersuchten deren Beine auf die darauf befindlichen Mikroorganismen. So konnten sie zeigen, daß besonders im Frühling auch diese Fliegen als Quellen für mikrobielle Destruenten fungieren.
Insgesamt werden hier wichtige Daten zum Kontext der Funktion von Ökosystemen vorgelegt. Verwesung/Zersetzung (engl. decomposition) ist eine fundamentale und alle terrestrischen Ökosysteme überspannende, mikrobielle Funktion. Und obgleich Pflanzenmaterial die dominierende Quelle organischer Materie darstellt, kann das Einfließen der Materialien aus Wirbeltierkadavern eine wichtige Ressource darstellen. Neben ihrer mikrobiologischen und biogeochemischen Grundlagenbedeutung sind die hier vorgestellten Erkenntnisse aber auch forensisch interessant: nach ausreichender Validierung und „Entschlackung“ solcher mikrobiologischen, metagenomischen Datensätze könnten sie zur relativ präzisen Bestimmung des PMI auch bereits stark verwester Leichname dienen und ggf. sogar Aussagen über den Ort des Versterbens (durch Untersuchung von Bodenproben) ermöglichen.



Kommentare (4)