Im zweiten Teil dieser Miniserie habe ich erklärt, warum die forensische RNA-Analyse eine hervorragende und überaus sinnvolle Ergänzung zur etablierten Standard-Analyse von DNA sein kann. In diesem letzten Teil will ich auf die überaus verschiedenen, vielfältigen Einsatzmöglichkeiten der forensischen RNA-Analyse eingehen, denn es ist besonders ihre Vielseitigkeit, die mich fasziniert.
Wenn man sich das Methodenrepertoire der forensischen Molekularbiologie einmal wie ein Schweizer-Armeemesser vorstellt, dann wäre die große Hauptklinge daran wohl gleichzusetzen mit der DNA-Analyse, also dem wichtigsten, hauptsächlich genutzten Instrument, das unverzichtbar ist und ohne das das Ganze einfach kein Schweizer-Armeemesser wäre. Aber dann gibt es eben auch diese vielen nützlichen und sehr verschiedenen Zusatzfunktionen, die in manchen Situationen unersetzlich sind: es gibt ja so Momente, da braucht man einfach nur einen Korkenzieher 😉 Und diese Zusatzfunktionen kann man sich als RNA-analytische Ansätze vorstellen, die zur Bearbeitung zusätzlicher und ebenso verschiedener forensischer Fragestellungen eingesetzt werden können, wie Schere, Korkenzieher, Kapselheber, Ahle, Feile und Zahnstocher die große Messerklinge ergänzen.
dann wäre die große Hauptklinge daran wohl gleichzusetzen mit der DNA-Analyse, also dem wichtigsten, hauptsächlich genutzten Instrument, das unverzichtbar ist und ohne das das Ganze einfach kein Schweizer-Armeemesser wäre. Aber dann gibt es eben auch diese vielen nützlichen und sehr verschiedenen Zusatzfunktionen, die in manchen Situationen unersetzlich sind: es gibt ja so Momente, da braucht man einfach nur einen Korkenzieher 😉 Und diese Zusatzfunktionen kann man sich als RNA-analytische Ansätze vorstellen, die zur Bearbeitung zusätzlicher und ebenso verschiedener forensischer Fragestellungen eingesetzt werden können, wie Schere, Korkenzieher, Kapselheber, Ahle, Feile und Zahnstocher die große Messerklinge ergänzen.
Die erste Erwähnung von RNA aus postmortalem Gewebe in der forensischen Literatur findet sich bereits im Jahr 1984 in einer deutschsprachigen Arbeit zur postmortalen Biosynthese von DNA und RNA [1]. Zehn Jahre später beschrieben Phang et al. die Durchführung einer Reverse-Transkription–PCR (RT-PCR) ausgehend von aus postmortalem Gewebe entnommener RNA [2]. Seit der Entwicklung der quantitativen PCR (s. auch hier), einer meiner persönlichen Lieblings- und Hauptforschungsmethoden, im Jahr 1996 [3], die eine exakte Bestimmung der Menge einer bestimmten (m)RNA in einer Probe ermöglicht und damit eine Aussage über ihren differentiellen Expressionsstatus erlaubt, ist das Interesse an der (m)RNA-Analytik und auch ihren forensischen bzw. rechtsmedizinischen Anwendungsmöglichkeiten deutlich angestiegen.
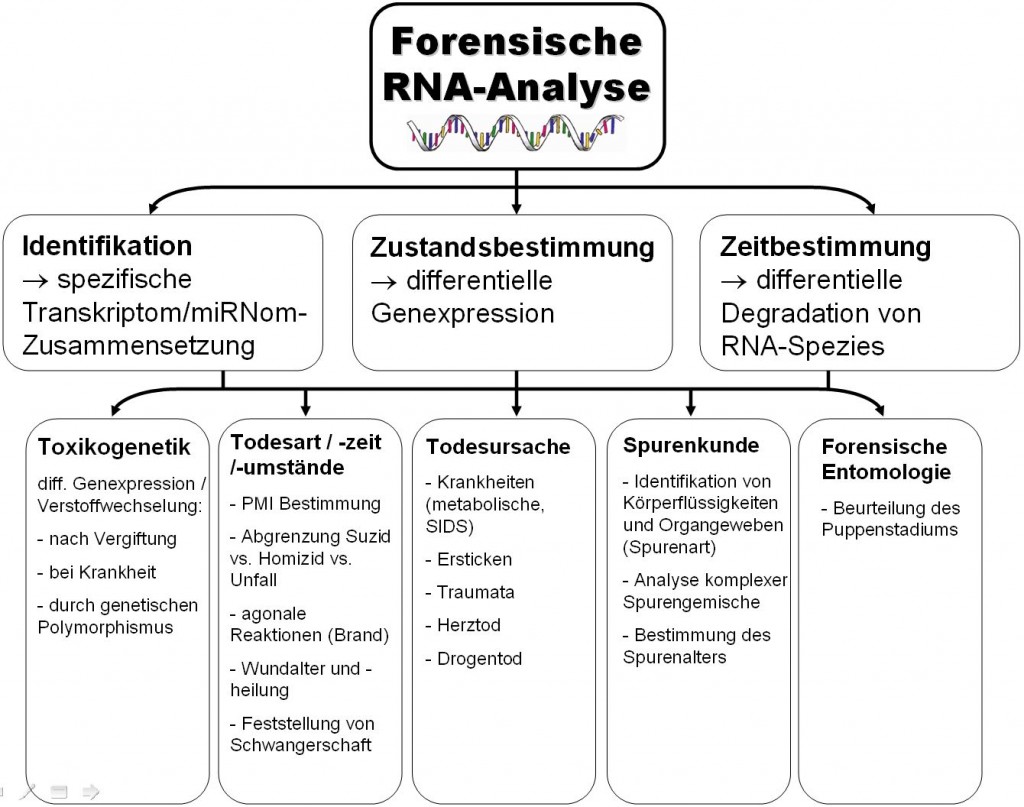
Zahlreiche Studien belegen inzwischen die überaus vielgestaltigen Möglichkeiten von Transkriptom- und Genexpressionsanalysen bei der Bearbeitung sehr verschiedener forensischer Fragestellungen:
Zu nennen wären hier vor allem Zustands- und Zeitbestimmungen [4], etwa die Abschätzung des post-mortem-Intervalls (PMI) und der Todeszeit [5-7], die zeitliche Eingrenzung von Wundalter und Heilungsprozessen [8-13], die Alterseinschätzung forensischen Spurenmaterials [14,15, s. auch hier] und des Puppenstadiums leichenbesiedelnder Insekten [16,17] sowie die post-mortale Feststellung von Schwangerschaften [18]. Aber auch molekulare Todesursachenermittlungen, z.B. zur Abgrenzung zwischen Suizid, Homizid und Unfalltod [19-25], zur Unterstützung der postmortalen Diagnostik rechtsmedizinisch relevanter Krankheitsbilder wie dem SIDS [26 bzw. hier] und toxikogenetische Untersuchungen können durch RNA-Analyse informiert werden [27-31].
Einige Forscher befassen sich darüber hinaus mit der Quantifizierung der Stabilität und Integrität von mRNA-Molekülen für die forensische Analytik [32]. RNA erwies sich dabei in mehreren Studien in postmortalen Geweben aber auch in teils sehr alten biologischen Spuren als deutlich haltbarer und weniger degradationsempfindlich, als zuvor gemeinhin angenommen [33, 34] und immer neue Quellen werden für eine RNA-Extraktion erschlossen, selbst aus Knochen ist inzwischen die Isolation prozessierbarer RNA gelungen [35].
Besonders wichtig und gut erforscht (u.a. im Rahmen meiner eigenen Forschungsbemühungen) ist aber der Einsatz der RNA-Analyse bei der Identifikation von Spurenarten [36-43], wofür sowohl die Untersuchung von mRNA als auch micro-RNA (miRNA) gut funktioniert (s. auch hier). Das Prinzip der Methode besteht darin, in biologischem Spurenmaterial spurenartspezifische, d.h. möglichst ausschließlich in den Zellen einer bestimmten Spurenart (z.B. Blut) transkribierte mRNAs oder miRNAs nachzuweisen und somit das Vorhandensein dieser Spurenart festzustellen. Vorteile dieses Verfahrens gegenüber den herkömmlichen meist (immun)chemischen Methoden zur Identifikation einer Spurenart ist die deutlich höhere Spezifität und die Möglichkeit, auch komplexe Mischungen von Spurenarten erfolgreich zu untersuchen.
Eine weitere Anwendung für die RNA-Analyse findet sich im von meinem Kollegen und mir begründeten Feld der „molekularen Ballistik“, die sich, wie regelmäßige LeserInnen dieses Blogs wissen werden, mit der molekulargenetischen Untersuchung nukleinsäurehaltiger Spuren befasst, welche durch Schusswaffeneinsatz gegen biologische Ziele entstehen und von äußeren und inneren Oberflächen der Waffen und/oder anderen Oberflächen (z.B. der Hand, die eine Schußwaffe abgefeuert hat) gesichert werden können.
Neben der Identität von Opfer(n), Täter(n) und anderen Beteiligten, die sich vermittels DNA-Analyse feststellen läßt, sind auch Erkenntnisse zum Hergang von Straftaten mit Feuerwaffeneinsatz von großem forensischem Interesse und genau hier kann die RNA-Analyse hilfreich sein. Bei Delikten mit Einsatz mehrerer Feuerwaffen kann es beispielsweise von entscheidender Bedeutung für die nachträgliche Rekonstruktion und Bewertung des Tatgeschehens sein, die verursachten Schusswunden den einzelnen beteiligten Schusswaffen zuordnen zu können. So ließe sich etwa ein Kopfschuss durch eine bestimmte Waffe nachweisen, indem durch den Schuss aus dem Wundkanal herausgeschleudertes Hirngewebe aus dem Inneren dieser Waffe detektiert werden könnte: die Gewebeherkunft einer biologischen Spur lässt sich anhand des Transkriptoms bzw. miRNoms bestimmen (s. auch hier).
Nachhausemitnehmbotschaft: Die forensische RNA-Analyse bereichert die forensische Molekularbiologie um eine große Vielfalt von ganz unterschiedlichen Einsatz- und Erkenntnismöglichkeiten, wofür die in diesem Artikel vorgestellten Anwendungen als – keineswegs erschöpfende – Belege gelten mögen. Sie kann dabei nicht nur die „traditionellen“ DNA-Untersuchungen ideal ergänzen, sondern auch in disziplinübergreifenden Ermittlungen eingesetzt werden und letztlich eigene Forschungsfelder begründen. Für die Zukunft gelangen im Zuge der Einführung und Verbreitung von NGS auch in forensischen Forschungsumgebungen Techniken wie Einzelzell-Transkriptomik und In-situ-RNA-Sequenzierung in Reichweite wodurch sich enorme Fortschritte und abermalige Erweiterungen der Möglichkeiten forensischer RNA-Analytik ergeben werden.
—
Ich selbst strebe an, mit diesen Entwicklungen Schritt zu halten (wir haben hier in Kiel gerade ein nagelneues NGS-Gerät bekommen 🙂 ) und neben der Bearbeitung innovativer Forschungsansätze (ich habe gerade eine DFG-Förderung zur Fortsetzung meines „molekulare Ballistik“-Projekts erhalten 🙂 \o/ ) auch der Akzeptanz und Validität RNA-basierter Verfahren in der rechtsmedizinischen und spurenkundlichen Routinearbeit beizuhelfen (wir hier in Kiel werden bald als erstes Labor in Deutschland die Akkreditierung gem. DIN/ISO 17025 für den Einsatz der forensischen RNA-Analytik zur Spurenartidentifikation erhalten 🙂 ).
____
Referenzen:
[1] M. Oehmichen, K. Zilles, [Postmortem DNA and RNA synthesis. Preliminary studies in human cadavers], Z. Rechtsmed. 91 (1984) 287-94.
[2] T. W. Phang, C. Y. Shi, J. N. Chia, C. N. Ong, Amplification of cDNA via RT-PCR using RNA extracted from postmortem tissues, J. Forensic Sci. 39 (1994) 1275-9.
[3] C. A. Heid, J. Stevens, K. J. Livak, P. M. Williams, Real time quantitative PCR, Genome Res. 6 (1996) 986-94.
[4] C. Courts, B. Madea, Ribonukleinsäure – Bedeutung in der forensischen Molekularbiologie, Rechtsmedizin 22 (2012) 135-44.
[5] M. Bauer, I. Gramlich, S. Polzin, D. Patzelt, Quantification of mRNA degradation as possible indicator of postmortem interval–a pilot study, Leg. Med. (Tokyo) 5 (2003) 220-7.
[6] A. Kimura, Y. Ishida, T. Hayashi, M. Nosaka, T. Kondo, Estimating time of death based on the biological clock, Int. J Legal Med 125 (2011) 385-91.
[7] S. T. Young, J. D. Wells, G. R. Hobbs, C. P. Bishop, Estimating postmortem interval using RNA degradation and morphological changes in tooth pulp, Forensic Sci Int. 229 (2013) 163-6.
[8] J. H. Sun, L. H. Nan, C. R. Gao, Y. Y. Wang, Validation of reference genes for estimating wound age in contused rat skeletal muscle by quantitative real-time PCR, Int. J Legal Med 126 (2012) 113-20.
[9] M. Takamiya, K. Saigusa, N. Nakayashiki, Y. Aoki, Studies on mRNA expression of basic fibroblast growth factor in wound healing for wound age determination, Int. J. Legal Med. 117 (2003) 46-50.
[10] M. Takamiya, K. Saigusa, R. Kumagai, N. Nakayashiki, Y. Aoki, Studies on mRNA expression of tissue-type plasminogen activator in bruises for wound age estimation, Int. J. Legal Med. 119 (2005) 16-21.
[11] Y. Sato, T. Ohshima, The expression of mRNA of proinflammatory cytokines during skin wound healing in mice: a preliminary study for forensic wound age estimation (II), Int. J. Legal Med. 113 (2000) 140-5.
[12] T. Ohshima, Y. Sato, Time-dependent expression of interleukin-10 (IL-10) mRNA during the early phase of skin wound healing as a possible indicator of wound vitality, Int. J. Legal Med. 111 (1998) 251-5.
[14] S. E. Anderson, G. R. Hobbs, C. P. Bishop, Multivariate analysis for estimating the age of a bloodstain, J Forensic Sci. 56 (2011) 186-93.
[15] C. Hampson, J. Louhelainen, S. McColl, An RNA expression method for agingforensic hair samples, J Forensic Sci. 56 (2011) 359-65.
[16] A. M. Tarone, D. R. Foran, Gene expression during blow fly development: improving the precision of age estimates in forensic entomology, J Forensic Sci. 56 Suppl 1 (2011) S112-S122.
[17] P. Boehme, P. Spahn, J. Amendt, R. Zehner, Differential gene expression during metamorphosis: a promising approach for age estimation of forensically important Calliphora vicina pupae (Diptera: Calliphoridae), Int. J Legal Med 127 (2013) 243-9.
[18] J. Gauvin, D. Zubakov, J. van Rhee-Binkhorst, A. Kloosterman, E. Steegers, M. Kayser, Forensic pregnancy diagnostics with placental mRNA markers, Int. J. Legal Med. 124 (2010) 13-7.
[19] Q. Wang, T. Ishikawa, T. Michiue, B. L. Zhu, D. W. Guan, H. Maeda, Intrapulmonary aquaporin-5 expression as a possible biomarker for discriminating smothering and choking from sudden cardiac death: A pilot study, Forensic Sci Int. 220 (2012) 154-7.
[20] D. Zhao, T. Ishikawa, L. Quan, T. Michiue, C. Yoshida, A. Komatu, et al., Postmortem mRNA quantification for investigation of infantile death: A comparison with adult cases, Leg. Med. 11 (2009) S286-S289.
[21] D. Zhao, B. L. Zhu, T. Ishikawa, D. R. Li, T. Michiue, H. Maeda, Quantitative RT-PCR assays of hypoxia-inducible factor-1alpha, erythropoietin and vascular endothelial growth factor mRNA transcripts in the kidneys with regard to the cause of death in medicolegal autopsy, Leg. Med (Tokyo) 8 (2006) 258-63.
[22] D. Zhao, T. Ishikawa, L. Quan, D. R. Li, T. Michiue, C. Yoshida, et al., Tissue-specific differences in mRNA quantification of glucose transporter 1 and vascular endothelial growth factor with special regard to death investigations of fatal injuries, Forensic Sci Int. 177 (2008) 176-83.
[23] B. L. Zhu, S. Tanaka, T. Ishikawa, D. Zhao, D. R. Li, T. Michiue, et al., Forensic pathological investigation of myocardial hypoxia-inducible factor-1 alpha, erythropoietin and vascular endothelial growth factor in cardiac death, Leg. Med (Tokyo) 10 (2008) 11-9.
[24] J. Becker, P. Schmidt, F. Musshoff, M. Fitzenreiter, B. Madea, MOR1 receptor mRNA expression in human brains of drug-related fatalities-a real-time PCR quantification, Forensic Sci Int. 140 (2004) 13-20.
[25] M. Iino, M. Nakatome, Y. Ogura, H. Fujimura, H. Kuroki, H. Inoue, et al., Real-time PCR quantitation of FE65 a beta-amyloid precursor protein-binding protein after traumatic brain injury in rats, Int. J Legal Med 117 (2003) 153-9.
[26] Courts, C., Grabmüller, M., & Madea, B. (2013). Dysregulation of heart and brain specific micro-RNA in sudden infant death syndrome. Forensic science international, 228(1), 70-74.
[27] K. A. Baken, R. J. Vandebriel, J. L. Pennings, J. C. Kleinjans, L. H. van, Toxicogenomics in the assessment of immunotoxicity, Methods 41 (2007) 132-41.
[28] G. Steiner, L. Suter, F. Boess, R. Gasser, M. C. de Vera, S. Albertini, et al., Discriminating different classes of toxicants by transcript profiling, Environ. Health Perspect. 112 (2004) 1236-48.
[29] M. J. Bartosiewicz, D. Jenkins, S. Penn, J. Emery, A. Buckpitt, Unique gene expression patterns in liver and kidney associated with exposure to chemical toxicants, J Pharmacol. Exp. Ther. 297 (2001) 895-905.
[30] S. J. Bulera, S. M. Eddy, E. Ferguson, T. A. Jatkoe, J. F. Reindel, M. R. Bleavins, et al., RNA expression in the early characterization of hepatotoxicants in Wistar rats by high-density DNA microarrays, Hepatology 33 (2001) 1239-58.
[31] H. K. Hamadeh, P. R. Bushel, S. Jayadev, K. Martin, O. DiSorbo, S. Sieber, et al., Gene expression analysis reveals chemical-specific profiles, Toxicol. Sci 67 (2002) 219-31.
[32] A. Koppelkamm, B. Vennemann, S. Lutz-Bonengel, T. Fracasso, M. Vennemann, RNA integrity in post-mortem samples: influencing parameters and implications on RT-qPCR assays, Int. J Legal Med 125 (2011) 573-80.
[33] D. Zubakov, M. Kokshoorn, A. Kloosterman, M. Kayser, New markers for old stains: stable mRNA markers for blood and saliva identification from up to 16-year-old stains, Int. J. Legal Med. 123 (2009) 71-4.
[34] H. Karlsson, C. Guthenberg, D. U. von, K. Kristenssson, Extraction of RNA from dried blood on filter papers after long-term storage, Clin. Chem. 49 (2003) 979-81.
[35] N. L. van Doorn, A. S. Wilson, E. Willerslev, M. T. Gilbert, Bone Marrow and Bone as a Source for Postmortem RNA*, J Forensic Sci. 56 (2011) 720-5.
[36] J. Juusola, J. Ballantyne, Messenger RNA profiling: a prototype method to supplant conventional methods for body fluid identification, Forensic Sci. Int. 135 (2003) 85-96.
[37] C. Haas, B. Klesser, C. Maake, W. Bar, A. Kratzer, mRNA profiling for body fluid identification by reverse transcription endpoint PCR and realtime PCR, Forensic Sci. Int. 3 (2009) 80-8.
[38] C. Haas, E. Hanson, A. Kratzer, W. Bar, J. Ballantyne, Selection of highly specific and sensitive mRNA biomarkers for the identification of blood, Forensic Sci Int. Genet. 5 (2011) 449-58.
[39] M. Visser, D. Zubakov, K. N. Ballantyne, M. Kayser, mRNA-based skin identification for forensic applications, Int. J Legal Med 125 (2011) 253-63.
[40] A. Lindenbergh, B. M. van den, R. J. Oostra, C. Cleypool, A. Bruggink, A. Kloosterman, et al., Development of a mRNA profiling multiplex for the inference of organ tissues, Int. J Legal Med 127 (2013) 891-900.
[41] A. Lindenbergh, P. Maaskant, T. Sijen, Implementation of RNA profiling in forensic casework, Forensic Sci Int. Genet. 7 (2013) 159-66.
[42] Sauer, E., Reinke, A. K., & Courts, C. (2016). Differentiation of five body fluids from forensic samples by expression analysis of four microRNAs using quantitative PCR. Forensic Science International: Genetics, 22, 89-99.
[43] Sauer, E., Extra, A., Cachée, P., & Courts, C. (2017). Identification of organ tissue types and skin from forensic samples by microRNA expression analysis. Forensic Science International: Genetics, 28, 99-110.



Kommentare (5)