Maus und Mensch haben auch ungefähr gleich viele Basenpaare, ca. 3 Milliarden, doch bei anderen Lebewesen ist so ein Vergleich komplizierter. Viele Insekten haben nur einige 100 Millionen Basenpaare, und das Bakterium E. coli hat gerade mal 4,5 Millionen. Archaeen wiederum haben nur etwa 1-3 Millionen Basenpaare. Um diese Lebewesen miteinander vergleichen zu können, müssen wir bei der Untersuchung der Genome die Gene nehmen, die in beiden zu vergleichenden Gruppen vorhanden sind.
Etwa die Hälfte der Gene von Archaea ist für sie einzigartig. Das heißt, dass z.T. nur eine halbe Million Basenpaare mit denen eines Menschen vergleichbar sind; und die können immer noch so verschieden sein, dass ein Vergleich völlig sinnlos wäre. Es gibt sie aber, die vergleichbaren Gene; aber es sind wenige und die Ähnlichkeit mit denen von Vertebraten schwanken verständlicherweise noch mehr als die zwischen Maus und Mensch. Gene für ganz bestimmte Enzyme finden sich z.B. in Archaeen, Bakterien, Küchenschaben und Menschen. Einige hundert Gene von Eukaryoten scheinen ihren Ursprung in den Archaeen zu haben. Und dann haben wir z.B. auch an die 40 Gene, die, soweit wir bisher wissen, nur bei Menschen und Bakterien auftauchen.
Der Grund dafür kann aber nicht nur die gemeinsame Abstammung sein, sondern ein (horizontaler) Gentransfer von den Bakterien zu uns. Horizontaler Gentransfer ist ein Problem in der Systematik, da die Gene, die wir zur Bestimmung der Verwandtschaft benutzen, sich in gewisser Weise erst später in das Genom eingeschlichen haben. Dieses Problem besteht in erster Linie bei den Prokaryoten und macht es den Forschern, die daran arbeiten, alles andere als leicht, eine Art Stammbaum aufzustellen. Prof. William Martin von der Universität Düsseldorf hat das Problem sehr schön illustriert:
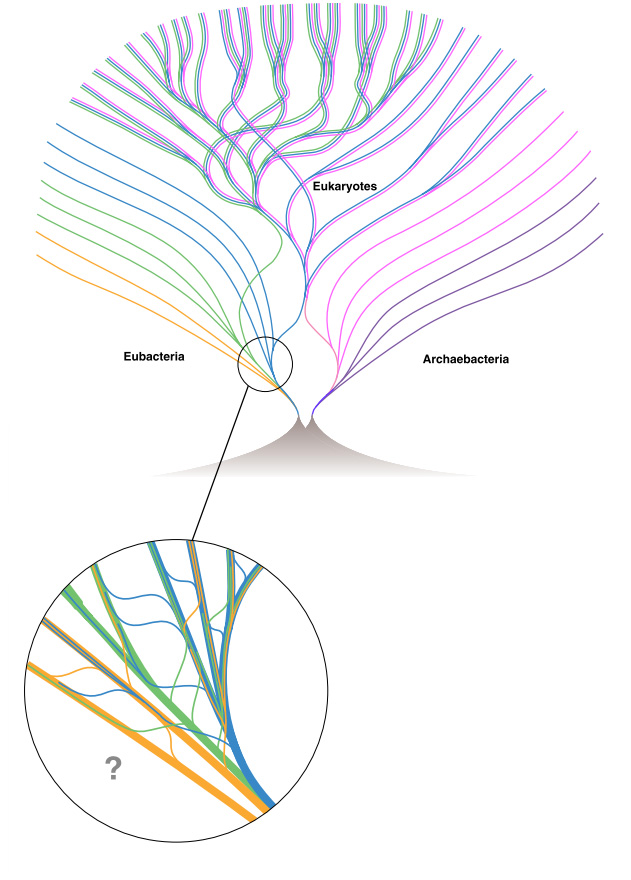
Vorschlag für einen Stammbaum des Lebens. Verschiedene Gruppen von Prokaryoten sind hier farblich unterschiedlich dargestellt. Die Abbildung soll das Problem des horizontalen Gentransfers in den Eubakterien beschreiben (verdeutlicht in der Vergrößerung unten links). (Quelle: verändert nach BioEssays 1999, 21:99-104 – Datei als PDF)
LUCA selbst könnte demnach sehr primitiv und frei von Organellen gewesen sein. Die Gene für die Speicher-Körnchen wären dann erst viel später durch Transfer in den drei Domänen verteilt worden.
Die Forscher der University of Illinois, die nun ihren Fund publizieren, widersprechen dieser Hypothese jedoch. Sie haben nämlich ein Enzym (eine Phosphatase) verfolgt, das in Bakterien, Archaeen und Eukaryoten vorkommt und eine wichtige Rolle bei Acidocalcisomen spielt, eben diesen in Archaea neu entdeckten Körnchen. Dort fördern diese Phosphatasen den Ionentransport in das Organell hinein. Ein Stammbaum aus beinahe 280 Organismen, basierend alleine auf diesem Enzym, ist fast identisch mit einem Stammbaum des Lebens, der auf hunderten von verschiedenen Genen basiert. Das würde bedeuten, dass die untersuchten Phosphate – und somit auch die Acidocalcisomen – sehr, sehr alt sind.
Etwa so alt wie LUCA.
Die Anwesenheit dieses Organells in Archaea und Bakterien und Eukaryoten lässt sich auf vielen Wege erklären, doch laut den Autoren ist die Erklärung, dass es in LUCA vorkam und nachträglich verloren ging, die statistisch wahrscheinlichste. Stammbäume werden auf dem Prinzip der Parsimonie aufgebaut, und danach ist die einfachste Form für einen Stammbaum auch die realistischste. Ihr Stammbaum zeigt, dass die Enzyme aus Acidocalcisomen zu Beginn da waren.
James Whitfield, einer der Autoren dieses Papers (und nebenbei bemerkt ein großartiger Banjo-Spieler) sagt dazu folgendes:
![]() Seufferheld, M., Kim, K., Whitfield, J., Valerio, A., & Caetano-Anolles, G. (2011). Evolution of vacuolar proton pyrophosphatase domains and volutin granules: clues into the early evolutionary origin of the acidocalcisome Biology Direct, 6 (1) DOI: 10.1186/1745-6150-6-50
Seufferheld, M., Kim, K., Whitfield, J., Valerio, A., & Caetano-Anolles, G. (2011). Evolution of vacuolar proton pyrophosphatase domains and volutin granules: clues into the early evolutionary origin of the acidocalcisome Biology Direct, 6 (1) DOI: 10.1186/1745-6150-6-50

Kommentare (12)