In der letzten Folge habe ich über Spurenkunde berichtet und erklärt, daß es wichtig ist, eine biologische Spur, aus der man DNA für die Herstellung eines DNA-Profils gewinnen möchte, genau zu charakterisieren.
In diesem Beitrag beschreibe ich, wie es gelingt, DNA aus den Zellen einer biologischen Spur zu isolieren. Wir streben dabei immer drei Ziele an: a) ausreichende Menge, b) ausreichende Integrität, c) ausreichende Sauberkeit.
a) Ist eine gewisse Mindestmenge an DNA nicht vorhanden, können wir kein solides und reproduzierbares DNA-Profil erstellen. Wir benötigen mindestens 50 pg DNA, um sicher zu sein. Ein Picogramm (pg) ist der tausendste Teil eines millionstel Milligramm – unvorstellbar wenig also und doch reicht die DNA in einer Spur manchmal nicht aus. Eine gute Aufreinigung muß eine möglichst hohe Ausbeute an DNA liefern, insbesondere, weil manche Spuren so klein sind, daß wir nur einen „Schuß” haben und alles auf eine Karte setzen müssen, um ausreichend DNA gewinnen zu können.
b) DNA ist zwar tough aber nicht unzerstörbar. Sie kann „kaputt” gehen, d.h. der Doppelstrang kann zerbrechen und ab einem bestimmten Fraktionierungsgrad taugt die DNA nicht mehr für die bei der Profilerstellung eingesetzte PCR-Amplifikation (warum erzähle ich später). Eine gute Aufreinigung muß möglichst intakte DNA liefern darf aber zumindest die DNA nicht noch weiter zerstören.
c) Manche Spuren sind extrem „siffig”, d.h. mit allen möglichen Umwelteinflüssen und/oder Chemikalien verunreinigt; man denke an Blut, das sich in einer Lache von Erbrochenem befindet oder auf einem mit psychedelischen Farben getünchten Jeansbeinkleid. In beiden Fällen kann die Spur mit Stoffen vermengt sein, sog. „Inhibitoren”, die die Effizienz oder Performance einer nachfolgenden chemischen Reaktion wie der PCR (s.o.) reduzieren, teils so stark, daß ein DNA-Profil gar nicht mehr gelingt. Eine gute Aufreinigung muß möglichst alle Inhibitoren aus der Präparation entfernen.
Es gibt eine Fülle an DNA-Isolationsmethoden und zig Firmen preisen ihr jeweiliges DNA-Extraktions-Kit – so nennt man eine gebrauchsfertige, praktische Zusammenstellung aller Chemikalien und sonstiger spezieller Vebrauchsmaterialien, die man für eine bestimmte Arbeit, z.B. eine DNA-Extraktion, benötigt (hier ein Beispiel) – als das beste an. Inzwischen gibt es auch zahlreiche, speziell auf forensischen Bedarf zugeschnittene Kits und Methoden.
Die Hersteller wissen natürlich, daß alle Methoden, die wir einsetzen, den drei oben genannten Ansprüchen genügen müssen und die meisten Kits leisten das auch. Sie unterscheiden sich daher vor allem in der grundlegenden Methode, der Bearbeitungszeit, der „Handhabbarkeit” und – ein wichtiger Faktor – dem Preis.
Man unterscheidet mehrere „grundlegende Methoden” denen jedoch allen gemein ist, daß das Ziel aller Extraktionsbemühungen immer ist, aus der extrem komplexen Mischung von Molekülen und Stoffen aus einer biologischen Spur, die nicht nur alle möglichen Verunreinigungen aus der Umwelt sondern auch für die angestrebte Analyse nutzlose oder gar beeinträchtigende Zellbestandteile enthält, möglichst ausschließlich DNA herauszufiltern.
Die folgende Tabelle stellt die bekanntesten Methoden vor:
Die organische 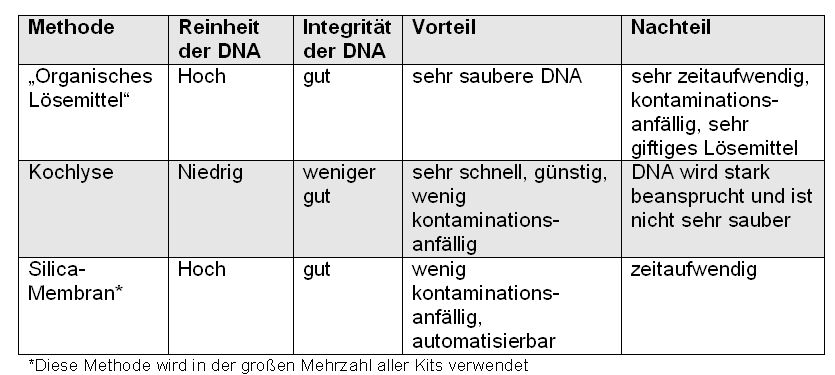 Methode beruht auf einer Phasentrennung (organische und wäßrige Phase) durch Zentrifugation mit der sehr wirksam DNA von Proteinen, Lipiden und sonstigem Zellschrott getrennt wird. Sie wird nicht mehr so oft (und gerne) verwendet, hauptsächlich, weil man dafür eine eigene Abzugshaube („Abzug”) benötigt, um überhaupt mit dem sehr giftigen und ätzenden Phenol und dem leicht flüchtigen Chloroform arbeiten zu dürfen. Zudem ist sie langwierig, durch zahlreiche Gefäßwechsel kontaminationsanfällig und nicht automatisierbar.
Methode beruht auf einer Phasentrennung (organische und wäßrige Phase) durch Zentrifugation mit der sehr wirksam DNA von Proteinen, Lipiden und sonstigem Zellschrott getrennt wird. Sie wird nicht mehr so oft (und gerne) verwendet, hauptsächlich, weil man dafür eine eigene Abzugshaube („Abzug”) benötigt, um überhaupt mit dem sehr giftigen und ätzenden Phenol und dem leicht flüchtigen Chloroform arbeiten zu dürfen. Zudem ist sie langwierig, durch zahlreiche Gefäßwechsel kontaminationsanfällig und nicht automatisierbar.
Die Kochlyse mit dem Chelatbildner Chelex® beruht auf darauf, die Zellen durch das Kochen der Extraktionsmischung „aufzuknacken” und dann mittels der Chelex-Chemikalie die zweiwertigen Kationen wegzuschnappen, die die DNA-zerstörenden Enzyme, die beim Aufknacken der Zellen freigesetzt werden, benötigen, um zu funktionieren. Die Methode geht durch das Kochen sehr „ruppig” mit der DNA um und ist eine „quick&dirty”-Technik, die man in der Analyse von möglicherweise empfindlichem Spurenmaterial eigentlich nicht einsetzt, um nicht Gefahr zu laufen, die DNA aus der Spur vollends unbrauchbar zu machen. Die Kochlyse findet aber noch regelmäßig Anwendung in der Abstammungsbegutachtung, wo man „frische” DNA aus einer Mundschleimhautprobe bearbeitet, für welche eine solche schnelle und brutale Extraktion recht gut geeignet ist. Zudem finden alle Schritte in einem einzigen Reaktionsgefäß statt und somit besteht durch das Entfallen von Transfers kaum Gefahr einer Kontamination. Man muß jedoch bedenken, daß die DNA nach der Kochlyse einzelsträngig vorliegt und daher ungeeignet für nachfolgende Analysen ist, die eine doppelsträngige DNA voraussetzen.
Die Silica-Membran-Methode ist die für Spurenuntersuchungen am besten geeignete Methode, da sie saubere DNA liefert, einen schonenden Umgang mit dem Material erlaubt und bei hohem Arbeitsaufkommen auch von einem Roboter durchgeführt werden kann. Sie beruht darauf, daß DNA in Gegenwart von hoch konzentrierten chaotropen Salzen reversibel an Silica (= Siliziumdioxid, SiO2) bindet. Die Zellen aus der Spur werden zunächst chemisch und durch Zugabe von Proteinase K, einem proteinspaltenden Enzym, aufgebrochen. Das Gemisch wird dann mit chaotropen Salzen versetzt und auf eine unbewegliche Silica-Membran gegeben, an die die DNA-Moleküle dann fest binden. Jetzt kann man den Zellschrott und sonstige Moleküle (unter Beibehaltung der Salzkonzentration) wegwaschen, bis am Ende nur noch die DNA an der Membran „klebt”. Indem man zum Schluß mit einer Lösung spült, die keine chaotropen Salze mehr enthält, löst sich die Bindung der DNA an der Membran, sie wird freigesetzt und ist nun schön sauber für die nächsten Schritte verfügbar. Fast alle kommerziell erhältlichen Kits verwenden eine Modifikation der Silica-Methode, die zum Teil sehr stark abgewandelt wird.
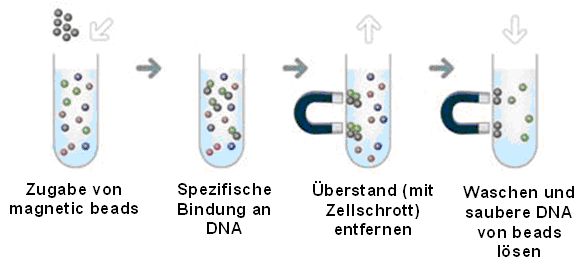
Ein Beispiel dafür sind Magnetkügelchen („magnetic beads”), die bei den forensischen Kits sehr beliebt sind, weil sie sehr saubere DNA hervorbringen.
Dabei werden winzige magnetische Kügelchen (braun) mit Silica oder einer anderen stark DNA bindenden Substanz (teilweise haben die Hersteller da ihr eigenes Geheimrezept) beschichtet. Im Verlauf der Extraktion binden dann die Kügelchen die DNA-Moleküle (grün). Dann wird das Extraktionsgefäß in ein starkes Magnetfeld gesetzt und die Kügelchen werden rein mechanisch im Gefäß festgehalten, während man den Zellschrott und anderes Material (rot und blau) aus der Präparation entfernen und danach mit sehr stringenten Bedingungen die DNA waschen kann. Am Schluß löst man chemisch die DNA wieder von den Kügelchen ab und trennt diese, wieder mit einem Magneten, von der sauberen DNA.
Manche Spuren bedürfen zusätzlich noch einer speziellen Anpassung und/oder Vorbehandlung für die Extraktion. Z.B. ist es oft nötig, vor der DNA-Extraktion aus Blutspuren die Erythrozyten zu entfernen, weil der Blutfarbstoff Hämoglobin ein bekannter PCR-Hemmstoff ist.
Ein anderer Problemfall sind Mischspuren z.B. aus Sperma und Vaginalsekret, wie sie oft bei der Spurensicherung nach Sexualdelikten auftreten. Solche Mischungen bestehen häufig aus 10 mal mehr Vaginalsekret als Sperma und daher besteht die Gefahr, daß das weibliche DNA-Profil (also das des Opfers) das männliche (das des Täters) „unterdrückt”, obwohl man ja gerade das des Täters unbedingt benötigt. Bei solchen Spuren bedarf es daher einer sogenannten „Differentiellen Lyse”. Hierfür macht man sich die unterschiedliche Stabilität von weiblichen und männlichen Spurenbestandteilen zunutze. Die weiblichen Zellen sind meist (Vaginal)Epithelzellen, die eine dünne und leicht zu brechende Zellmembran aufweisen, wohingegen die männlichen Zellen Spermien sind, die eine recht derbe, stabile Hülle haben. Man wählt zu Beginn der Extraktion Lysebedingungen (mit SDS und Proteinase K), die nur die Epithelzellen aufbrechen, nicht aber die Spermien. Aus dem Gemisch entfernt man dann die Nicht-Spermien-DNA und setzt anschließend härtere Lysebedingungen ein (wie oben aber zusätzlich noch DTT) , die selbst die Spermien aufknacken können. Danach kann man aus dem Gemisch gezielt die Spermien-DNA isolieren, die nun, ohne Überlagerung von weiblicher DNA, analysiert werden kann.
Am Ende des Extraktionsprozesses sollte man also ausreichende, halbwegs intakte und saubere DNA haben, die man dann als Grundlage für die Erstellung eines DNA-Profils einsetzen kann. SOLLTE. Leider läuft aber bei der Extraktion, ganz besonders bei schwierigem Spurenmaterial, nicht immer alles glatt und es kann passieren, daß nicht genug DNA gewonnen wurde oder die extrahierte DNA noch immer mit Inhibitoren verunreinigt ist. Man sollte also, bevor es weitergeht, herausfinden, wieviel DNA man denn nun hat und ob sie ausreichend sauber und intakt ist. Und wie das geht, erfahren Sie im nächsten Teil unserer beliebten Serie 😉



Kommentare (26)