Heute erscheint die aktuelle Ausgabe der Zeitschrift BIOspektrum (Springer Verlag) mit einem Special zu PCR/qPCR. Für dieses Special habe ich einen Artikel geschrieben, der exclusiv für die Leser von blooD’N’Acid auch hier zu lesen ist 🙂 (Als hilfreich für die Lektüre könnten sich die vorhergehenden Beiträge zu PCR, DNA-Quantifizierung und STR-Multiplex-PCR erweisen. )
___________________
Der Einzug der Untersuchung von Nukleinsäuremolekülen in die forensischen Wissenschaften hat sich seit den 1980er Jahren als großer Erfolg und unersetzliche Bereicherung für Rechtsprechung und Rechtspflege erwiesen und bildet bis heute das Hauptbetätigungsfeld der forensischen Molekularbiologie [1]. Eine besonders wichtige Aufgabe unserer Disziplin besteht in der Erstellung von DNA-Profilen. Ein DNA-Profil ist eine Auflistung genetischer Merkmale, die in ihrer Kombination individualspezifisch, d.h. einzigartig für die Person sind, von der die DNA stammt. Man verwendet solche Profile, um unkenntliche Leichen zu identifizieren, einen Täter der Beteiligung an einem Verbrechen zu überführen oder um ein biologisches Abstammungsverhältnis zwischen zwei Personen, etwa eine Vaterschaft, nachzuweisen.
Aber auch bei der RNA-basierten Interpretation von komplizierten Spurenmustern, wie sie häufig an Tatorten von Verbrechen vorgefunden werden, kann die forensische Molekularbiologie einen entscheidenden Beitrag leisten [2]. Indem sie durch die Identifikation der vorhandenen Spurenarten und die Aufschlüsselung komplexer Mischspuren die Interpretation des Spurenbildes und seiner Entstehung ermöglicht, kann eine auf objektiven Belegen begründete Rekonstruktion des Tathergangs erfolgen.
Zur Erfüllung aller dieser Aufgaben sind die Polymerasekettenreaktion (PCR) und verschiedene spezialisierte Abwandlungen dieses Verfahrens von zentraler Bedeutung und die PCR ist aus der modernen Forensik nicht mehr wegzudenken.
Quantitative PCR zur Bestimmung der Menge und Reinheit von Spuren-DNA
Die Analyse biologischer Spuren von Tatorten stellt für forensische Biologen häufig eine Herausforderung dar. Nicht selten bestehen solche Spuren aus nur einem einzelnen telogenen Haar oder einigen wenigen Hautschuppen, die ein Täter hinterlassen hat. Zum sehr geringen DNA-Gehalt solcher Spuren kommt noch erschwerend hinzu, daß die DNA darin durch Umwelteinflüsse wie Feuchtigkeit oder UV-Strahlung beschädigt und/oder durch Stoffe aus der Umgebung (Inhibitoren) verunreinigt sein kann. Sowohl beschädigte als auch mit Inhibitoren verunreinigte DNA kann in schwer oder nicht interpretierbaren DNA-Profilen resultieren. Aus diesem Grund muß vor der eigentlichen DNA-Profilerstellung die Integrität, Menge und mögliche Verunreinigung der aus den Spuren extrahierten DNA analysiert werden. Hierzu eignet sich besonders gut ein quantitatives PCR-Verfahren. Während der zyklenweise Ablauf der Standard-PCR wie eine „black box“ ist, in die man nicht hineinsehen kann und an deren Ende man lediglich eine bestimmte Menge des gewünschten Produktes erhält, ermöglicht die quantitative PCR (qPCR) [3] eine fluoreszenzbasierte exakte Messung des Zuwachses des PCR-Produktes in jedem Zyklus der PCR.
Zusätzlich zu den für die PCR üblichen Primern benötigt dieses Verfahren dafür sogenannte Sonden: kurze Nukleotidsequenzen, die komplementär für genau den von den beiden Primern flankierten DNA-Abschnitt und zusätzlich mit einem Fluoreszenzfarbstoff markiert sind. In der Annealingphase der qPCR lagern sich nicht nur die Primer sondern auch diese fluoreszenzmarkierten Sonden an ihre jeweilige Zielsequenz an. Wenn dann die DNA-Polymerase, die am 3’-Ende jedes Primers ansetzt, um eine Kopie des Gegenstrangs zu erzeugen, in ihrem Fortschreiten auf die Sonde trifft, verdrängt sie die Sonde von ihrer Bindestelle und zerstört sie dabei. Hierdurch wird der in der intakten Sonde inaktivierte Fluoreszenzfarbstoff freigesetzt und durch einen im qPCR-Gerät verbauten Laser zum Leuchten angeregt, was von einem Sensor detektiert wird. Es wird dabei pro PCR-Zyklus desto mehr Farbstoff freigesetzt, je mehr Sonden binden und zerstört werden können. Die kontinuierlich ansteigende Fluoreszenzsignalstärke, die somit direkt mit dem Zuwachs an PCR-Produkt korreliert ist und in jedem Zyklus erneut gemessen wird, ermöglicht in der Folge einen genauen Rückschluss auf die Ausgangskonzentration der DNA zu Beginn der qPCR. So läßt sich also bestimmen, wie viel DNA aus dem Spurenmaterial gewonnen wurde und ob es ausreichend für nachfolgende Untersuchungen ist.
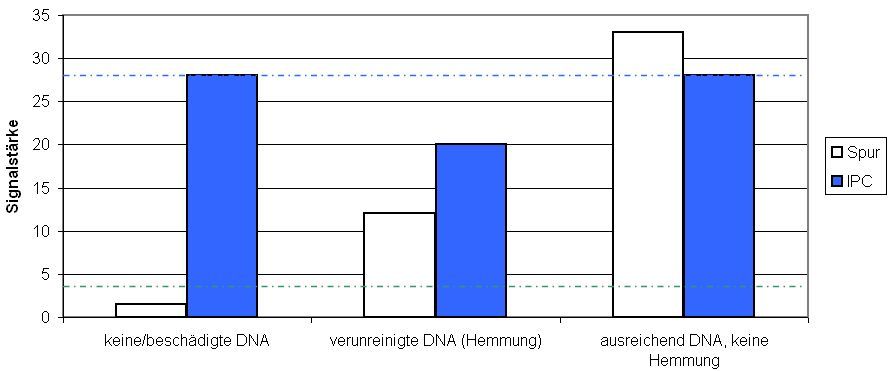
Um auch eine mögliche Verunreinigung mit Inhibitoren entdecken zu können, setzt man jedem Reaktionsgefäß mit einem DNA-Extrakt aus Spurenmaterial noch eine künstliche DNA als interne Positivkontrolle (IPC) zu und führt sie durch alle Schritte unter gleichen Bedingungen mit. Für die Fluoreszenzsignalstärke der IPC am Ende der qPCR gibt es einen genauen und vordefinierten Soll-Wert. Wird dieser nicht erreicht, ist der Reaktionsansatz sehr wahrscheinlich mit Hemmstoffen verunreinigt, die den normalen Ablauf der PCR gestört haben und sich für nachfolgende Analysen mit der Spuren-DNA, z.B. der STR-Multiplex-PCR, als problematisch erweisen können (Abb.1).
STR-Multiplex-PCR zur Erzeugung von DNA-Profilen
Ein besonders aufwendiges und komplexes PCR-Verfahren stellt die STR-Multiplex-PCR dar. Dieses Verfahren ermöglicht die gleichzeitige Vervielfältigung von üblicherweise 17 und mehr „short tandem repeat“(STR)-Systemen und ist die Grundlage für die Erstellung eines individuellen DNA-Profils. Ein STR-System ist ein definierter DNA-Abschnitt, der eine Reihe von kurzen, hintereinander angeordneten Wiederholungen eines nur wenige Basen umfassenden DNA-Motivs umfasst, z.B. AGGA. STR-Systeme liegen in der sogenannten Microsatelliten-DNA, sind über das ganze Genom verteilt ist und jeder Mensch besitzt zwei Kopien, auch als „Allel“ bezeichnet, eines jeden STR-Systems. Diese eignen sich deshalb hervorragend zur Unterscheidung von Personen, weil jede bestimmte Kombination der Anzahlen von Motivwiederholungen in einer Gruppe von acht oder mehr STR-Systemen mit extrem hoher Wahrscheinlichkeit nur in einem einzigen Menschen vorkommt [4].
Um nicht 17 und mehr PCR-Ansätze für alle zu vervielfältigenden STR-Systeme vorlegen zu müssen, wofür ohnehin meist nicht genug DNA-Extrakt zur Verfügung steht, werden alle Ansätze zusammengeführt und zu einer einzigen Multiplex-Reaktion vereinigt. Die Feinabstimmung der Reaktionsbedingungen und biochemischen Parameter für eine PCR, in der 17 und mehr Primerpaare untergebracht werden müssen und die mit möglichst ähnlicher Amplifikationsseffizienz und ohne sich dabei gegenseitig zu behindern funktionieren sollen, stellt eine höchst komplexe, aufwendige Prozedur dar.
Viele forensische Labore verwenden daher kommerziell erhältliche, gebrauchsfertige Zusammenstellungen, sogenannte „Kits“, für die Durchführung von STR-Multiplex-PCR. Solche Kits haben den Vorteil, daß sie sehr zeit- und arbeitsparend eingesetzt werden können, da sie den äußerst hohen gesetzlichen Ansprüchen an die Methodenvalidierung forensischer Labore genügen. Die Primer, die in solchen Kits enthalten sind, sind mit einem von mehreren verschiedenfarbigen Fluoreszenzfarbstoffen markiert. Dies ermöglicht nach Fertigstellung der STR-Multiplex-PCR die Sortierung der dabei entstandenen PCR-Produkte in einem Elektropherogramm: eine Voraussetzung für die Erstellung eines eindeutigen DNA-Profils.
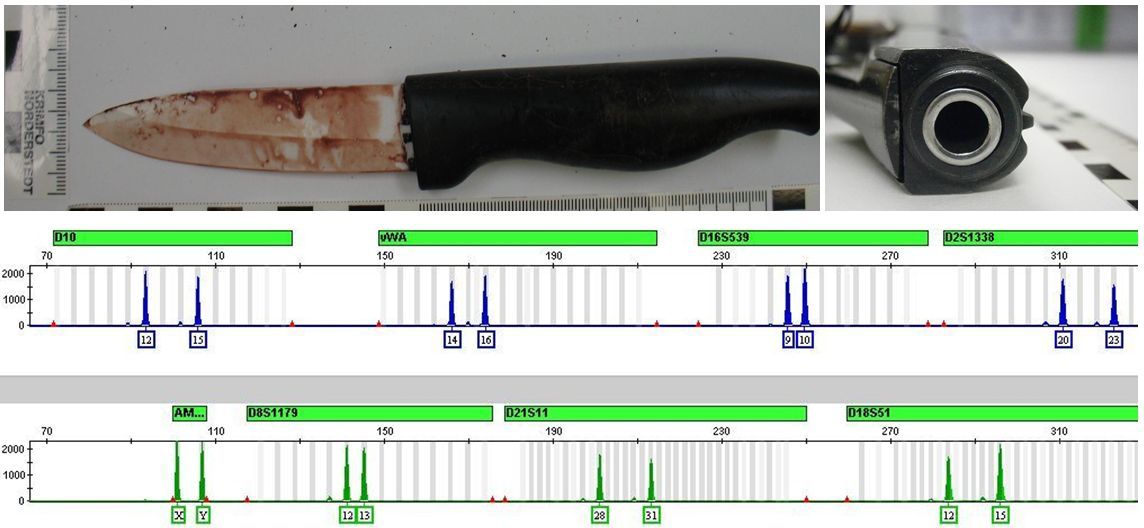
Die Vervielfältigung von STR-Systemen mittels STR-Multiplex-PCR ist derzeit die bedeutendste und weltweit am stärksten verbreitete Methode des DNA-profilings. Außerdem basieren heute alle von Polizeien und anderen Ermittlungsbehörden unterhaltenen DNA-Datenbanken, in denen die Profile von überführten Verbrechern aber auch unbekannten Tatortspuren gespeichert werden, auf STR-basierten DNA-Profilen. Die STR-Multiplex-PCR hat sich bewährt: sie ist ein robustes und verlässliches Verfahren und ermöglicht ein profiling auch noch aus sehr geringen DNA-Mengen von 100 Picogramm. Es ist uns damit vor kurzem sogar gelungen, aus der DNA, die aus dem Inneren von Schußwaffenläufen gewonnen wurde [5], die DNA-Profile der durch diese Waffen getöteten Personen zu erstellen (Abb. 2).
Reverse-Transkription-PCR zur RNA-basierten Spurenartidentifikation
Seit kurzem wird auch die Analyse der Ribonukleinsäure (RNA) in der forensischen Fallarbeit verwendet. Die RNA, genauer die mRNA, ist das direkte molekulare Korrelat der Genexpression und eine quantitative Analyse des momentaufnahmenartigen Bestands der RNA-Moleküle in den Zellen aus biologischem Spurenmaterial, das sogenannte „Transkriptom“, lässt die genaue Bestimmung der Zusammensetzung selbst schwieriger Mischspuren zu [6]. Das ist insbesondere bei der Rekonstruktion von Sexualdelikten von entscheidender Wichtigkeit, da hier Mischspuren aus bis zu fünf oder sechs verschiedenen Spurenarten auftreten können. Zu diesem Zweck müssen mittels PCR bestimmte für eine Spurenart spezifische mRNA-Moleküle nachgewiesen werden.
Die PCR benötigt jedoch grundsätzlich DNA als Ausgangsmaterial. Deshalb muß die aus dem Spurenmaterial gewonnene RNA zunächst in komplementäre DNA (cDNA) umgewandelt oder transkribiert werden. Im normalen „Zellalltag“ verläuft die Transkription aber in die andere Richtung, es wird also DNA zu RNA transkribiert. Aus diesem Grund bezeichnet man die Umschrift von RNA zu cDNA als umgekehrte oder „reverse“ Transkription (RT) und das Enzym, das sie durchführt ist die „Reverse Transkriptase“. Die RT ist dabei nicht einmal annährend so effizient wie die PCR und erzeugt deshalb vergleichsweise große Varianzen im RT-PCR-Verfahren. Wichtiger als die absolute Effizienz der RT ist jedoch, daß sie die Mengenverhältnisse der mRNAs aus der Ursprungspopulation, dem RNA-Extrakt, exakt im Reaktionsprodukt, der cDNA, abbilden kann. Der Nachweis spurenartspezifischer mRNAs wird also indirekt über den Nachweis komplementärer, spurenartspezifischer cDNAs geführt. An die RT schließt sich dann eine PCR zum Nachweis dieser cDNAs an. Dabei handelt es sich entweder um eine qPCR mit fluoreszierenden Sonden (s.o.), wodurch die gesuchten cDNAs in „Echtzeit“ detektiert werden können. Oder man führt eine Standard-PCR mit fluoreszenzmarkierten Primern durch, ganz ähnlich dem STR-Multiplex-Verfahren (s.o.), an die dann die kapillarelektrophoretische Detektion und Identifikation der PCR-Produkte und Darstellung im Elektropherogramm angeschlossen wird (Abb. 3).
Literatur
1. Jeffreys A J, Wilson V, Thein S L (1985). Individual-specific ‘fingerprints’ of human DNA. Nature 316: 76-79.
2. Courts C, Madea B (2012). Ribonukleinsäure – Bedeutung in der forensischen Molekularbiologie. Rechtsmedizin 22: 135-144.
3. Gibson U E, Heid C A, Williams P M (1996). A novel method for real time quantitative RT-PCR. Genome Res. 6: 995-1001.
4. Butler J M (2009). Fundamentals of Forensic DNA Typing. Academic Press.
5. Schyma C, Madea B, Courts C (2013). Persistence of biological traces in gun barrels after fatal contact shots. Forensic Sci Int. Genet. 7: 22-27.
6. Juusola J, Ballantyne J (2005). Multiplex mRNA profiling for the identification of body fluids. Forensic Sci. Int. 152: 1-12.




Kommentare (5)