Schon wieder ein RNA-Artikel? Jawohl. Nicht nur bin ich bekanntlich Fan von RNA im Allgemeinen und miRNA im Speziellen, ich fühle mich auch angesichts eines meiner eigenen Forschungsschwerpunkte (forensische RNA-Analytik) aufgefordert, dem vielfach anzutreffenden DNA-Chauvinismus in der (forensischen) Genetik entgegenzutreten (vielleicht hätte ich das Blog haiR’N’Acid nennen sollen…) und immer wieder die RNA ins Zentrum des Interesses zu rücken.
So auch diesmal, da ich von der Möglichkeit, RNA als therapeutisches Mittel, als Medikament einzusetzen, berichten will. Um zu verstehen zu können, worum es gehen wird, sollte man mit dem Konzept der RNAi vertraut sein und/oder zur Vorbereitung nochmal hier vorbeilesen.
Mit Medikamenten, deren Wirkmechanismus auf RNAi beruht, macht man sich endogene, posttranskriptionale Genregulationswege für therapeutische Zwecke zunutze. Das Ziel besteht darin, die Expression von Genen, die auf irgendeine Weise an der zu bekämpfenden Krankheit beteiligt sind, herunterzuregulieren oder ganz auszuschalten. Die Auslöser oder „Trigger“ der RNAi sind dabei typischerweise kurze, doppelsträngige RNAs (dsRNAs), deren einer Strang komplementär zu einem Teil der mRNA-Sequenz ist, die für das zu regulierende Genprodukt kodiert, wodurch diese mRNA und das korrespondierende Protein reduziert bzw. eliminiert wird. Solche dsRNAs kann man auf verschiedene Weisen von außen zuführen, als synthetische Oligonukleotide oder als DNA-Matrizen, von denen die RNAi-Trigger dann in den Zielzellen erst noch transkribiert werden müssen (ganz ähnlich wie bei miRNAs, die ja die endogenen also körpereigenen Trigger für RNAi sind), das wird vektor-basierte transkriptionale RNAi genannt.
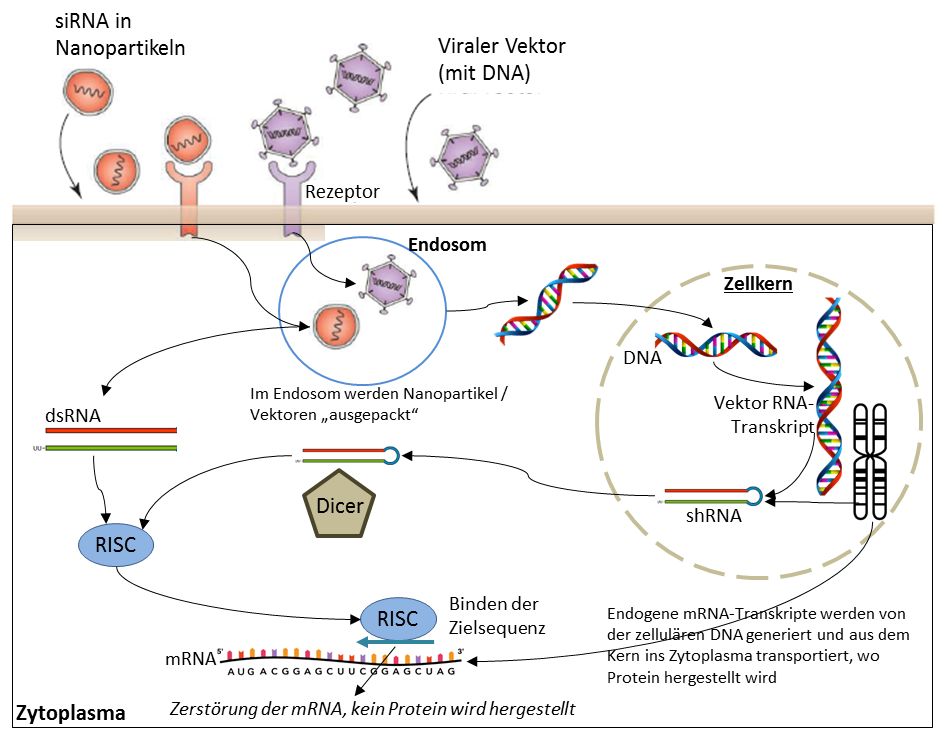
Die zelluläre Aufnahme erfolgt non-spezifisch oder über rezeptorvermittelte Endocytose. Nanopartikel entladen ihre dsRNA-Fracht direkt ins Zytoplasma. Der entsprechende Trigger tritt dann, je nach Art des Transports, in den RNAi-Signalweg entweder beim RISC oder schon bei Dicer ein. Von Transkriptionsvorlagen werden shRNAs transkribiert, die sogar schon im Zellkern in den RNAi-Signalweg eintreten. In jedem Fall entsteht am Ende ein aktiver RISC-Komplex, der das zugehörige mRNA-Ziel findet und zerstört. / RISC: RNA-induced silencing complex; shRNA: small hairpin RNA
Zentral für die therapeutische Nutzbarkeit von RNAi-Triggern ist es, sie in genau die Zellen des Körpers hineinzubekommen, in denen sie wirken sollen (Tropismus). Für diesen Transport werden hauptsächlich drei verschiedene Techniken genutzt: Nanopartikel, einfache Konjugate und virale Vektoren. Mit einigen dieser Technologien ließen sich bereits in menschlichen Lebern Gene sehr langanhaltend stillegen, so daß monatliche oder gar nur vierteljährliche Medikamentengaben erreichbar scheinen [1,2].
Dieses Jahr wurden auch die ersten Phase-III Klinischen Studien zu RNAi-Medikamenten (ALN-TTR02 und ALN-TTRsc) zur Behandlung der familiären Amyloidpolyneuropathie beendet, die die Expression des mutierten krankheitsverursachenden Transthyretin-Gens (TTR) verhindern. Es gibt darüber hinaus noch eine Reihe anderer möglicher Ziele für RNAi-Medikamente in der Leber, z.B. bei Hepatitis B, verschiedenen häufigen metabolischen und kardiovaskulären Erkrankungen aber auch selteneren Leiden wie primärer Hyperoxalurie u.a. Die Leber ist allerdings vergleichsweise leicht „anzusteuern“, weil sie gemäß ihrer Funktion ohnehin Partikel aus dem Blutkreislauf entfernt und es ist weniger klar, wie gut sich andere Gewebe durch solche Wirkstoffe ansteuern lassen.
Ein anderes Problem bei RNAi-Medikamenten kann darin bestehen, daß die externen RNAi-Trigger die zelleigene Maschinerie (s. Abbildung oben) zur Prozessierung von RNAs verwenden und diese sozusagen verstopfen können, so daß die körpereigenen RNAs (z.B. miRNAs) für RNAi nicht mehr in ausreichendem Maß bearbeitet und fertiggestellt werden können. Das gilt vor allem für externe RNAi-Trigger, die von DNA-Vorlagen mit starken Promotoren transkribiert werden und kann durchaus schädliche Effekte haben [3]. Durch Verbesserungen im Design von modernen RNAi-Triggern, die eine effizientere Prozessierung ermöglichen, kann man aber die Last für die Prozessierungsmaschinerie deutlich verringern [4].
Die Entwicklungsgeschichte von RNAi-Therapeutika war übrigens ganz schon ruckelig: am Anfang, so zwischen 2002 und 2005 interessierten sich nur kleine Biotech-Firmen mit Focus auf Oligonukleotid-Therapeutika dafür. Dann gab es 2006 den Nobelpreis für die Entdeckung der RNAi und es entbrannte plötzlich ein riesiger Run in der Pharmabranche auf die Technologie [5], der gekennzeichnet war durch Bieterkriege und völlig überzogene Erwartungen, bis es 2008 zu einem erheblichen Einbruch und Rückschlag kam, der bis 2011 andauerte und so heftig war, daß lange unklar war, ob sich die Industrie davon würde erholen können. Erst nach eindeutigen Nachweisen für die Stillegung von Zielgenen im menschlichen Organismus gewann das Feld Glaubwürdigkeit zurück und begann die Industrie wieder zu wachsen und in der ersten Hälfte von 2014 wurde bereits wieder eine Milliarde Dollar investiert [6] und das Interesse hält an.
Ich finde RNA-basierte Medikamente extrem spannend und vielversprechend und neben klassischer RNAi wird ja inzwischen auch mit RNAse-H-Antisense-Technologie als therapeutischer Ansatz gearbeitet (zum Vergleich der Methoden s. [7]). Sie haben das Potential, sehr viel präziser und verträglicher zu sein als andere Therapieformen, da sie sehr genau, gerade in der Krebstherapie, wo man gezielt mutierte Genprodukte ausschalten will, auf den Patienten und seine genetische Ausstattung zugeschnitten werden und noch dazu und im Gegensatz zu beispielsweise Antikörperen, sehr leicht und günstig hergestellt werden können, ganz analog zu CRISPR also, das ja auch auf RNA-Basenpaarung (gRNA), basiert.
______
Referenzen:
[1] Coelho, T., Adams, D., Silva, A., Lozeron, P., Hawkins, P. N., Mant, T., … & Munisamy, M. (2013). Safety and efficacy of RNAi therapy for transthyretin amyloidosis. New England Journal of Medicine, 369(9), 819-829.
[2] Wooddell, C. I., Rozema, D. B., Hossbach, M., John, M., Hamilton, H. L., Chu, Q., … & Deckert, J. (2013). Hepatocyte-targeted RNAi therapeutics for the treatment of chronic hepatitis B virus infection. Molecular Therapy, 21(5), 973-985.
[3] Grimm, D., Streetz, K. L., Jopling, C. L., Storm, T. A., Pandey, K., Davis, C. R., … & Kay, M. A. (2006). Fatality in mice due to oversaturation of cellular microRNA/short hairpin RNA pathways. nature, 441(7092), 537-541.
[4] Gu, S., Jin, L., Zhang, Y., Huang, Y., Zhang, F., Valdmanis, P. N., & Kay, M. A. (2012). The loop position of shRNAs and pre-miRNAs is critical for the accuracy of dicer processing in vivo. Cell, 151(4), 900-911.
[5] Haussecker, D. (2012). The business of RNAi therapeutics in 2012. Molecular Therapy-Nucleic Acids, 1.
[6] Maraganore, J. (2014). Technology: Investment suggests RNAi is on the up. Nature, 510(7503), 35-35.
[7] Vickers, T. A., Koo, S., Bennett, C. F., Crooke, S. T., Dean, N. M., & Baker, B. F. (2003). Efficient reduction of target RNAs by small interfering RNA and RNase H-dependent antisense agents A comparative analysis. Journal of Biological Chemistry, 278(9), 7108-7118.



Kommentare (1)