Eines meiner Forschungsprojekte, das ich zusammen mit einem geschätzten Kollegen (Rechtsmediziner und Wundballistiker) aus Bern und einer meiner beiden Doktorandinnen bearbeite, befasst sich mit der Entstehung von Spuren nach Schußwaffengebrauch. Seit letztem Jahr wird dieses Projekt zur molekularen Ballistik von der DFG gefördert und ich habe hier auch schon einige Male darüber berichtet (die verlinkten Artikel können auch zur Vorbereitung auf bzw. zum besseren Verständnis des folgenden Berichts dienlich sein).![]()
Bisher hatten sich unsere Anstrengungen nur auf die DNA aus dem Waffeninneren konzentriert. Wir konnten zeigen, daß Opfer-DNA durch Backspatter ins Innere von Schußwaffen gelangen und dort erhalten bleiben, auch Nachschüsse und lange Lagerzeiten überstehen und die Waffe sozusagen ein DNA-Gedächtnis ausbilden kann: sie „erinnert“ sich, wer mit ihr erschossen wurde. Diese Erkenntnisse konnten wir inzwischen sogar schon bei den Ermittlungen zu einem prominenten Mehrfachmord zur Anwendung bringen.
Wer sich aber an meinen anderen Forschungsschwerpunkt erinnert, wird nicht verwundert sein, daß ich mich irgendwann zu fragen begann, ob die Spuren im Waffeninneren nicht auch noch auswertbare RNA enthalten könnten. Zusammen mit einem hoch motivierten Medizin-Doktoranden (ja, die gibt es) gingen wir dieser Frage nach und vor kurzem wurde nun eine weitere Publikation zur Veröffentlichung angenommen [1]: wir beschreiben darin, wie wir erstmalig RNA (mRNA und micro-RNA (miRNA)) aus dem Backspatter im Inneren von Schußwaffen untersuchten und stellen nun eine Möglichkeit vor, anhand der RNA-Zusammensetzung im Spurenmaterial Rückschlüsse auf die Eintrittstelle des Projektils (Kopfschuß oder kein Kopfschuß) zu ziehen. Der Clou ist, daß wir für diese Arbeit die Methoden zur Extraktion von DNA und RNA integriert haben und so nicht nur die RNA sondern auch die DNA aus der Spur isolieren konnten: es geht also keine Information verloren.
Die Studie bestand im Prinzip aus drei Schritten oder Phasen:
Phase I: Um einen Kopfschuß von einem Schuß an eine andere Stelle zu unterscheiden, stellten wir die Hypothese auf, daß bei einem Kopfschuß neben Blut auch Hirngewebe aus dem Einschußdefekt per Backspatter herausgeschleudert wird. Um diese Hypothese zu testen, mußten wir Blut von Mischungen aus Blut und Hirngewebe unterscheiden können. Dazu brauchten wir RNA-Kandidaten, die besonders viel in Hirngewebe vorhanden (= exprimiert) sind. Also nahmen wir uns diverse Datenbanken und die Literatur vor und suchten solange, bis wir drei vielversprechende Kandidaten, die mRNA „C1orf61“ und die beiden miRNAs miR-124a und miR-124*, gefunden hatten.
Wir fertigten dann Proben aus Blut (B), Hirngewebe (TL) und Mischungen aus Blut und Hirngewebe (M) an und untersuchten mittels quantitativer PCR (qPCR), wie stark oder schwach unsere RNA-Kandidaten in den unterschiedlichen Probenarten exprimiert waren.
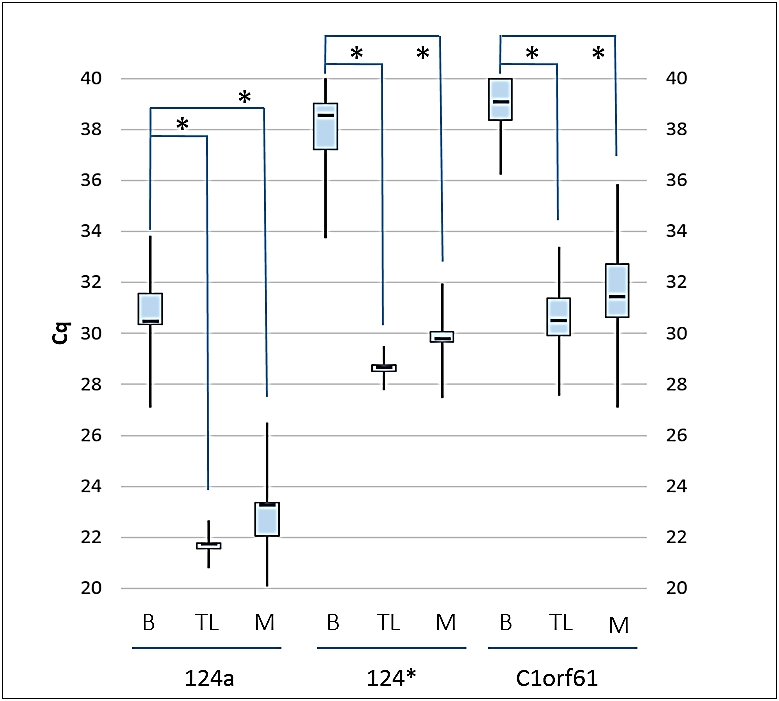
Cq: Cycle of quantitation (je höher, desto schwächer ist die RNA exprimiert); B: Blut, TL: Hirngewebe; M: Mischung aus Blut und Hirngewebe; *: signifikanter Unterschied
Die Abbildung zeigt die Ergebnisse aus Phase 1. Man erkennt sehr schön, daß alle drei RNA-Kandidaten (X-Achse) ausgezeichnet zwischen dem Vorhandensein und Fehlen von Hirngewebe unterscheiden können. Diese Art der Darstellung nennt man Box-Plot und sie gestattet, eine Zusammenfassung mehrerer Meßwerte im Vergleich zwischen verschiedenen Gruppen. Die „Box“ zeigt dabei die Streuung der Werte an, der horizontale Strich in der Mitte ist der Mittelwert und die „Antennen“, die oben und unten aus der Box herauskommen, entsprechen in unserem Fall der dreifachen Standardabweichung (3SD). Wichtig ist nun, festzustellen, daß sich bei keinem der RNA Kandidaten die Bereiche Mittelwert ± 3SD zwischen Proben mit und ohne Hirn überlappen. Das bedeutet, daß die Wahrscheinlichkeit, einen gemessenen Cq-Wert falsch zu interpretieren, unter 1% liegt (angedeutet durch *). Wenn ich also für z.B. miR-124a in einer unbekannten Probe einen Cq-Wert von 22 messe und daraufhin auf das Vorhandensein von Hirngewebe in der Probe schließe, dann ist die Wahrscheinlichkeit, daß in dieser Probe in Wirklichkeit kein Hirngewebe vorhanden ist, kleiner als 1%. Wir waren also mit der Unterscheidungskraft unserer RNA-Kandidaten zufrieden und entschlossen uns, alle drei für die kommenden Schritte zu verwenden.



Kommentare (7)