Ich habe noch nicht sehr viel über Epigenetik geschrieben, dabei ist das Thema höchst interessant. Epigenetik ist inzwischen längst auch für die forensische Genetik von Bedeutung und mein Team und ich werkeln gerade an einem ersten Forschungsansatz. Es ist also höchste Zeit, in einem Basics-Artikel ein wenig von den Grundlagen der Epigenetik zu erklären. (Zur Vorbereitung der Lektüre dieses Artikels empfiehlt sich bei Bedarf die Auffrischung der Kenntnisse über DNA). Das Wort „Epigenetik“ mit seiner griechischen Vorsilbe „Epi“ (für „um …herum“ oder „zusätzlich zu“) bezeichnet Vorgänge, Phänomene und Prozesse, die als zusätzlich zur „klassischen“ Genetik aufgefasst werden können, wenn man unter letzterer einmal die Kodierung von Aminosäuren in der Abfolge (=Sequenz) der Nukleotide der DNA und die Vererbung nach den Mendelschen Regeln versteht.
Epigenetische Veränderungen betreffen daher nicht die Nukleotidsequenz, also den eigentlichen Inhalt des Genoms, sondern z.B. die Entscheidung, ob und/oder in welcher Reihenfolge bestimmte Passagen des Genoms abgelesen, ob und wann also ein Gen exprimiert wird oder nicht. Sie können so erheblichen Einfluss auf die Funktion eines Gens und das Erscheinungsbild (Phänotyp) einer Zelle ausüben und sie sie können auch auf Tochterzellen und möglicherweise sogar Abkömmlinge eines Organismus vererbt werden.
Ich habe schon einen Basics-Artikel zur Regulation der Genexpression geschrieben, durch Nachlesen dessen man die Notwendigkeit und Bedeutung der differentiellen Genaktivität noch einmal rekapitulieren kann. Noch einmal ganz kurz: da (bis auf wenige Ausnahmen) jede Zelle das gesamte Erbgut eines mehrzelligen Organismus enthält, von dieser riesigen Informationsmenge jedoch nur einen Bruchteil für die Ausübung ihrer eigentlichen Funktion benötigt, muß eine menschliche Zelle (durch innere und äußere Einflüsse) genau steuern, welche der über 20.000 Gene zu welchem Zeitpunkt ausgeprägt (= exprimiert) werden, um sie mit genau den Genprodukten zu versorgen, die sie in jedem Moment gerade benötigt. Die zur Verfügung stehenden Steuerungsmechanismen der Genexpression sind vielfältig und kompliziert und epigenetische Veränderungen gehören dazu.
Wenn wir noch einmal die Restaurantanalogie aus jenem Basics-Artikel hernehmen und uns die DNA wie ein Kochbuch vorstellen mögen, dann entsprächen genetische Veränderungen (= Mutationen) also Änderungen im Text der Rezepte, wohingegen epigenetische Veränderungen eher an den Rand gekritzelten Anmerkungen und Büroklammern entsprächen, die hier und da Seiten zusammenhalten, so daß sie nicht aufgeschlagen und abgelesen werden können. Man kann sich leicht vorstellen, daß beide Arten von Veränderungen einen große Auswirkung auf das spätere Ergebnis haben können, ob nun in einem Rezept „Zucker“ durch „Salz“ ersetzt wird (Mutation) oder ein Gala-Dinner nur aus Suppe und Nachtisch ohne Hauptgang besteht (epigenetische Veränderung).
Nun gibt es ganz verschiedene Mechanismen zur epigenetischen Regulation und man kann sogar die Interferenz bei der Genexpression durch kleine RNAs (RNAi), zu denen auch mein Liebling, die miRNA, gehört, dazu zählen. Neben RNAi sind besonders wichtige Mechanismen aber die Methylierung von DNA-Nukleotiden und die Modifikation von Histon-Proteinen.
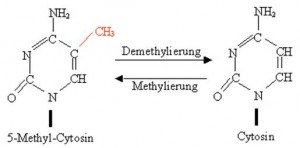 Methylierung bedeutet das chemische Anfügen einer Methylgruppe, -CH3 (s. Abbildung ). Epigenetische Veränderungen in Form von DNA-Methylierungen betreffen dabei immer nur Cytosin-Nukleotide und besonders dann, wenn diese in Sequenzumgebungen vorkommen, die reich an der Nukleotidabfolge Cytosin-Guanin (CG) sind, die sogenannten „CpG-Inseln“. Die Anfügung der Methylgruppe übernimmt dabei das Enzym DNA-Methyltransferase (DNMT). Aber wie kommt man nun von methylierten Cytosinen zur Steuerung der Genregulation?
Methylierung bedeutet das chemische Anfügen einer Methylgruppe, -CH3 (s. Abbildung ). Epigenetische Veränderungen in Form von DNA-Methylierungen betreffen dabei immer nur Cytosin-Nukleotide und besonders dann, wenn diese in Sequenzumgebungen vorkommen, die reich an der Nukleotidabfolge Cytosin-Guanin (CG) sind, die sogenannten „CpG-Inseln“. Die Anfügung der Methylgruppe übernimmt dabei das Enzym DNA-Methyltransferase (DNMT). Aber wie kommt man nun von methylierten Cytosinen zur Steuerung der Genregulation?
Man kann sich so eine Methylierung wie eine Markierung oder (etwas bildlicher) wie diese Kunststoffstacheln, die manche Gebäude zur Abwehr von Tauben auf den Dachfirsten angebracht haben, vorstellen. Zur Genregulation werden nämlich Cytosine methyliert, die in einer Promotorregion eines Gens liegen. Der Promotor ist so etwas wie der Startblock für die Transkription, an dem sich die RNA-Polymerase und alle sonst noch nötigen Proteine (z.B. Transkriptionsfaktoren) treffen und für die Transkription bereit machen. Wenn man sich die RNA-Polymerase nun wie eine Taube vorstellt, die sich gemütlich auf der DNA niederlassen möchte, dann wird sie von den „Methylgruppenstacheln“ auf den Cytosinen daran gehindert: sie kann sich nicht setzen und fliegt wieder weg; es gibt keine Transkription und das Gen wird nicht aktiviert. Methylierte Cytosine in Promotorregionen sind also (fast immer) Markierungen für inaktivierte Gene.
Man darf sich eine Promotormethylierung aber nicht als irreversible Abschaltung eines Gens vorstellen. Neuere Erkenntnisse belegen, daß es sich dabei vielmehr um einen überaus dynamischen Prozess handelt, der in verschiedenen Organismen zudem völlig unterschiedlich eingesetzt wird. Bakterien nutzen sie z.B., um ihre DNA zu schützen: Bakterien stellen Enzyme her, die DNA zerstören können, um sich gegen Fremd-DNA, z.B. aus Phagen („bakterienfressende“ Viren), zu schützen. Damit nicht auch ihre eigene DNA diesen Enzymen zum Opfer fällt, wird sie methyliert, denn auch diese Enzyme (sind wie Tauben und) können sich nicht auf methylierter DNA niederlassen. Wichtig ist auch zu erwähnen, daß Methylierungsmuster vererbt werden können. Bei der Zellteilung, im Rahmen derer die DNA der Ausgangszelle kopiert (repliziert) wird, kann eine DNMT das Methylierungsmuster der Eltern-DNA auf die neu gebildete Tochter-DNA übertragen.
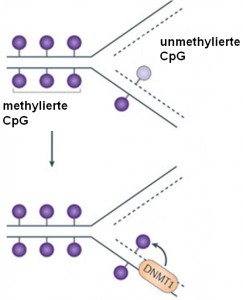
oben: der neu gebildete DNA-Strang (gestrichelte Linie) ist noch unmethyliert
unten: anhand der Methylierung am alten Strang (durchgehende Linie) erkennt die DNMT, wo eine neue Methylierungsmarkierung hin muß und fügt sie an der richtigen Stelle an
So werden neben der eigentlichen DNA-Information auch die Markierungen also die „Anmerkungen“ zum Ablesen an neue Zellen weitergegeben, wodurch sichergestellt wird, daß sie den gleichen Genregulationszustand erhalten.
Diese Art der epigenetischen Steuerung, die einer Zelle die genaue Bedienung ihres Bedarf an Genprodukten ermöglicht, spielt aber nicht nur in adulten Zellen eine wichtige Rolle, sondern auch und schon in den frühen Entwicklungsphasen eines Organismus: bis auf einige Ausnahmen, bei denen eine elternspezifische (maternale oder paternale) Ausprägung einer genetischen Anlage schon in der Keimzelle angelegt wird (sog. „Imprinting“), werden in der befruchteten Eizelle (Zygote) alle epigenetischen Markierungen auf der DNA ausradiert. Durch während der Embryonalentwicklung neu etablierte Markierungen kann dann gesteuert werden, welche Zellen sich wie entwickeln, man sagt „differenzieren“, z.B. ob eine frühe pluripotente Zelle zu einer Leber- oder Hirnzelle wird. In dieser Phase kann die Methylierung bestimmter Gene erheblichen Einfluss auf den heranwachsenden Organismus ausüben und z.B. möglicherweise die sexuelle Orientierung beeinflussen.
Einige Forscher, darunter der gestern mit der Otto-Warburg-Medaille ausgezeichnete R. Jaenisch, arbeiten u.a. daran, bereits differenzierte, also „erwachsene“ Zellen durch Eingriffe in u.a. die epigenetische Regulation wieder auf den Stand sogenannter induzierter, pluripotenter Stammzellen (iPSC) zurückzusetzen und dann zu Zellen für die Modellierung menschlicher Erkrankungen z.B. M. Parkinson, umzuprogrammieren.
Wenig überraschend ist fehlgesteuerte epigenetische Regulation inzwischen längst in Zusammenhang mit zahlreichen Erkrankungen gebracht worden. Diese umfassen nicht nur „klassische“ Imprinting-Defekte wie das Prader-Willi– oder das Angelmann-Syndrom, sondern auch Krebs, wo z.B. durch Hypermethylierung Tumor-Suppressorgene stillgelegt werden können, aber auch Herzkrankheiten, geistige Erkrankungen und viele weitere. Einige Forscher schätzen die Rolle der Epigenetik in Krankheit und Gesundheit für die Zukunft inzwischen sogar schon als größer ein, als die der „klassischen“ Genetik.
Zur enormen Bedeutung der Epigenetik passt auch, daß seit den frühen 2000er Jahren bekannt ist, daß das Verhalten eines Individuums direkten Einfluss auf die epigenetischen Markierungen seiner DNA hat. Man weiß, daß z.B. die Ernährung oder Luftverschmutzung die Veränderung von Methylierungsmustern bewirken können. Gewissermaßen wird also unser Genom und die Regulierung unserer Gene von der Umwelt beeinflusst.
Und als wäre das nicht schon erstaunlich genug, wird seit einiger Zeit vermutet, daß epigenetische Markierungen, die ein Organismus im Laufe seines Lebens erworben hat, sogar irgendwie an seine Nachkommen weitergegeben werden können. Wenn das wirklich stimmt, entspräche das gewissermaßen einer teilweisen post-mortalen Rehabilitation der Ideen von Lamarck, der dachte, daß von Organismen erworbene Eigenschaften auf deren Nachkommen übertragen werden könnten! So wurde z.B. beobachtet, daß eine Mäusen künstlich antrainierte Angst vor dem Geruch von Acetophenon auch bei deren Nachkommen und sogar deren Enkeln (wenn auch schwächer) noch auftritt. Aber auch in Menschen fand man solche Effekte, z.B. hatten Männer, die vor der Pubertät Hunger gelitten hatten, weniger häufig Enkelsöhne mit Herzerkrankungen oder Diabetes als Männer, die immer ausreichend ernährt waren. Eine andere Studie zeigte, daß Männer, die vor dem Alter von 11 Jahren zu rauchen begonnen hatten, häufiger Söhne mit Übergewicht hatten, als die entsprechende Kontrollgruppe.
Besonders interessant und erforderlich für eine echte transgenerationale Vererbung wären aber Veränderungen, die sich auch noch in der vierten Generation, also nicht nur in den Kindern und Enkeln sondern auch den Urenkeln nachweisen lassen. M. Skinner behauptet, einen solchen Effekt in einem Mausmodell beobachtet zu haben und sieht bereits „einen der größten Paradigmenwechsel in der Wissenschaft in den letzten Jahren“ heraufziehen.
Allerdings gab es Probleme bei der Replikation seiner Daten und eine Arbeit seiner Gruppe mußte wegen Datenfälschung zurückgezogen werden. Viele Wissenschaftler sind und bleiben daher skeptisch hinsichtlich der Vererbung epigenetischer Markierungen auf nachfolgende Generationen. Wir haben jedenfalls bis heute keine gute Idee, wie genau ein solcher Übergang stattfinden könnte, wie z.B. solche Markierungen die große Auslöschung in der Zygote (s.o.) überstehen sollen. Das heißt aber keineswegs, daß es nicht möglich ist und das Forschungsfeld ist und bleibt daher ungeheuer spannend.
Eine andere wichtige Form der epigenetischen Regulation stellt die Histon-Modifikation dar. Histone sind Proteine, die an die DNA binden und eine Organisation und bei Bedarf Verpackung des Erbgutes ermöglichen. Es sind fünf Haupt-Histon-Proteine bekannt H1, H2A, H2B, H3 und H4. Je 2 H2A und H2B bilden mit je 2 H3 und H4 achtfache Proteinkomplexe, um die sich die DNA in ca. zwei großen linksgängigen Windungen legen kann. Das H1 fungiert als eine Art Gerüst für den DNA-Doppelstrang und spielt eine Rolle bei der Transkription. Zusammen mit der DNA bilden die Histone das sogenannte Nukleosom:
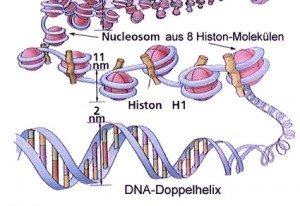
DNA windet sich um Histonkomplexe und bildet Nukleosomen
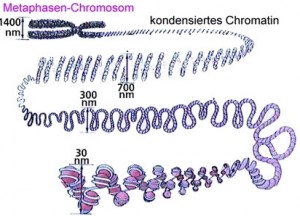
Die gesamte DNA ist in regelmäßigen Abständen (beim Menschen ca. 50 – 60 bp) in Nukleosomen organisiert. Diesen Zustand, diesen Komplex aus DNA und Proteinen bezeichnet man als Chromatin. Das Chromatin kann bei Bedarf, z.B. wenn die DNA für die Zellteilung/Mitose verpackt werden muß, extrem stark bis zur höchsten Stufe des Metaphasenchromosoms kondensiert werden:
Die DNA muß aber, wenn sich die Zelle nicht gerade teilt, jederzeit für verschiedene Vorgänge wie Transkription und Replikation zugänglich für die ausführenden Enzyme sein, da unter anderem der Doppelstrang aufgetrennt und die Einzelstränge abgelesen werden müssen. Dieser Zugang kann nun durch die Histonproteine, um die die DNA gewunden ist, modifziert werden: die Enden oder Schwänze der Histonproteine ragen aus den Histonkomplexen heraus und können mit chemischen Modifikationen versehen werden. Das können, ganz ähnlich wie schon für die DNA beschrieben, Methylierungen sein, häufiger aber sind es Acetylierungen (= Anbringen einer Acetylgruppe) und seltener Phosphorylierungen (= Anbringen einer Phosphatgruppe) und noch einige weitere (s. Abbildung).
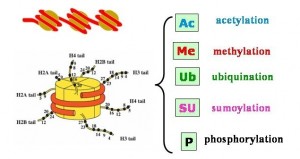
oben: DNA ist um Histonkomplexe gewunden
unten: aus den Histonen ragen mehrere Schwänze (tail) nach ausßen, an deren Aminosäuren (schwarze Punkte) verschiedene Modifikationen angebracht werden können
Die Modifikationen haben, abhängig von der Stelle (= der Aminosäure in der Histonsequenz), an der sie angebracht werden, unterschiedliche Auswirkungen. Acetylierungen etwa können von den Histon-Acetyltransferasen (HAT) nur an Lysin-Aminosäuren angebracht werden. Dort verringern sie die Wechselwirkung des Lysins mit der DNA wodurch letztlich die Chromatinstruktur aufgelockert und die DNA zugänglich für Transkription etc. wird. Histon-Methylierungen hingegen können neben Lysin auch an Arginin angefügt werden und zwar nicht nur einzeln, sondern bis zu drei Stück und können so ganz verschiedene Auwirkungen haben, von der Aktivierung bis zur Stillegung eines Gens.
Eine andere Theorie besagt, daß die Histon-Modifikationen nicht die Wechselwirkung zwischen Histon und DNA beeinflussen, sondern statt dessen als Bindestellen für Proteine dienen, die wiederum die Chromatinstruktur beeinflussen können.
Verschiedene Histon-Modifikationen wirken jedenfalls auf verschiedene Weisen; eine Acetylierung an der einen Stelle hat wahrscheinlich andere Auswirkungen als eine Acetylierung an einer anderen Stelle. Zudem können mehrere Modifikationen zur gleichen Zeit auftreten und dann zusammen wirken und das Verhalten des Nucleosoms verändern. Das noch unbestätigte Konzept, demzufolge mehrere dynamische Histon-Modifikationen die Genexpression auf systematische und reproduzierbare Weise regulieren, z.B. durch „Ablesen“ der Modifikationen durch entsprechende Proteine, wird daher auch als „Histon-Code“ bezeichnet.
Klar ist: durch die Modifikationen an den Histonen kann die Chromatin-Struktur gewissermaßen modelliert und der Zugang zu verschiedenen Bereichen der DNA sehr genau reguliert werden. Auch Histon-Modifikationen werden daher bei der DNA-Replikation erhalten. Bei der Zellteilung können Enzyme die Modifikationen der Histone im Eltern-DNA-Strang auf die Histone der neu gebildeten Tochter-DNA übertragen.
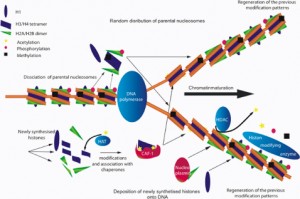
Während der DNA-Replikation werden die Nucleosomen aus dem Elternstrang zufällig auf die Tochterstränge verteilt, daher müssen die Histonmodifikationen des Elternstrangs sofort in den Tochtersträngen nachgebaut werden
Zusammen mit der Methylierung der DNA stellen Histon-Modifikationen der Zelle ein wichtiges und überaus komplexes Instrumentarium zur Steuerung zentraler Vorgänge zur Verfügung, welches nur einen Teil der Möglichkeiten epigenetischer Regulation (zu der ja auch RNAi zu zählen ist) darstellt. Seit kurzem erst weiß man übrigens auch, daß Histon-Modifizierung und DNA-Methylierung keineswegs unabhängig voneinander sind. Die Verständigung zwischen beiden Prozessen wird durch die Interaktion sog. SET-domain-Histon-Methyltransferasen und DNA-Methyltransferasen ermöglicht und ist für einen normalen Ablauf von Entwicklungsprozessen erforderlich, ist aber auch von Bedeutung bei der Umprogrammierung von normalen Zellen zu Krebszellen.
Neuere Studien belegten kürzlich außerdem eine Rolle von Histon-Modifikationen beim alternativen Spleißen. Epigenetische Modifikationen bestimmen damit also nicht nur, welche Teile des Genoms wann exprimiert werden, sondern beinflussen auch die kombinatorische Vielfalt in der sie zu den mannigfaltigen Funktionsträgern der Zelle zusammengefügt werden.
_____
Nachtrag am 07.05.2014: Ein umfangreiches Review zum Thema transgenerationaler Vererbung epigenetischer Markierungen wurde kürzlich bei Cell veröffentlicht. Es trägt den Untertitel “Myths and Mechanisms” und kommt zum Schluß, daß nach wie vor unklar ist, in welchem Umfang diese Vererbung auftritt und durch die Umwelt beeinflusst wird.



Kommentare (85)