
Rechter Oberschenkelknochen mit teilweise eröffneter Markhöhle und fehlender proximaler Epiphyse; aus [1]

Vier lose Zähne, die bei der Exhumierung gefunden wurden; Wurzeln intakt, Kronen deutlich abgenutzt; aus [1]
- bei allen Arbeitsschritten wurden Einmal-Ganzkörperschutzanzüge, Mundschutz, Einmal-Handschuhe, und Schutzbrillen getragen
- Arbeitsplätze, -flächen und –geräte wurden mit chlorbleichehaltigen Reinigungsmitteln geputzt und mit UV-Licht bestrahlt; dies entspricht einer chemischen und physikalischen Zerstörung von DNA
- die DNA-Extraktion aus dem Knochenmaterial erfolgte in einem speziellen Laborbereich, der für Niedrigmengen an DNA ausgewiesen und besonders sauber und geschützt ist
- die PCR-Amplifikation erfolgte in einem eigenen, räumlich von den anderen Arbeiten getrennten Bereich
- es wurden Positiv- und Negativkontrollen über alle Arbeitsschritte hinweg mitgeführt; besonders Negativkontrollen, also Reaktionsansätze, denen keine Knochen-DNA zugesetzt wird und die kein Signal erzeugen darf, sind wichtig, um mögliche Kontaminationen zu entdecken
- es erfolgten mehrfache PCR-Amplifikationen derselben Probe, um Ergebnisse zu bestätitgen bzw. Artefakte zu erkennen und zu verwerfen
Außerdem wurden die Knochenteile stets getrennt von anderen Proben aufbewahrt und eine strenge Gewahrsamskette (chain-of-custody) eingehalten. Die Arbeiten an dem Knochenmaterial wurden durch eine Frau durchgeführt, so daß eine Kontamination durch y-chromosomale DNA der bearbeitenden Person ausgeschlossen war.
Die Oberflächen der Knochenteile wurde mit einem Dremel-Rotationswerkzeug abgeschliffen. Der Kieferknochen erwies sich als zu instabil und wurde an dieser Stelle zurückgestellt und nicht für die DNA-Extraktion (s.u.) verwendet. Die Schienbein- und Oberschenkelknochen wurden dann mit einer Sektionssäge in Teile zerlegt (s. Photos). Einzelne Segmente und die Zähne wurden weiter dekontaminiert, indem sie erst für ca. 15 min in Chlorbleiche inkubiert, dann mit Reinstwasser abgespült und schließlich kurz in Ethanol getaucht wurden. Die so behandelten Stücke wurden über Nacht an einem sterilen Ort getrocknet. Am Folgetag wurden die Stücke einzeln in einer speziellen Knochenmühle („SPEX 6750 Freezer Mill“) unter flüssigen Stickstoff (um keine DNA-schädigende Hitze entstehen zu lassen) zu feinem Pulver zermahlen.
Zur Extraktion der DNA aus dem Knochenpulver setzte das Labor gleich drei Methoden parallel ein, um möglichen Schwierigkeiten durch das hohe Alter der Knochen zu begegnen: 1. eine Methode, die Loreille et al. zuvor für die DNA-Exktraktion aus einer mumifizierten Leiche beschrieben hatten [2], 2. eine Methode auf Basis von Silica-Säulen und 3. eine organische Extraktion (die Beschreibungen der 2. und 3. Methode habe ich in einem älteren Artikel schon gegeben). Die Quantifizierung der Knochen-DNA wurde mittels quantitativer PCR durchgeführt (wie das geht, beschrieb ich hier).
Im Anschluss wurden sowohl 16 autosomale als auch 17 y-chromosomale STR- Systeme umfassende Profile aus der Knochen-DNA erstellt. Dazu wurde die DNA mit verschiedenen Kits amplifiziert und die entstandenen Produkte kapillarelektrophoretisch aufgetrennt. Im Gegensatz zu autosomalen sind Y-chromosomale STR-Profile Haplotypen, so wie die mtDNA, und daher nicht so aussagekräftig, wie die autosomalen (also auf den Chromosomen 1-22 liegenden) STR-Systeme, eignen sich aber sehr gut, um väterliche Erblinien zurückzuverfolgen.
Das Labor erhielt auch Vergleichsspeichelproben möglicher lebender Verwandter von Zeke Harper, mit denen auf gleiche Weise verfahren wurde, die aber erst bearbeitet wurden, nachdem die Bearbeitung der Knochenproben vollständig abgeschlossen war. Auch dies diente der Vermeidung möglicher Kontaminationen.
Ergebnisse:
Autosomale STR-Systeme:
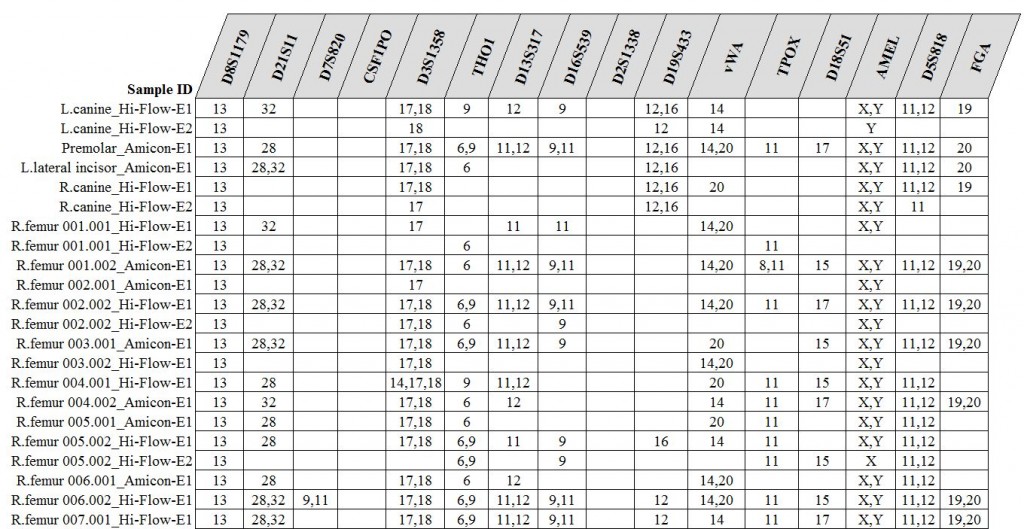
Zeilen: einzelne Proben, jeweils mit Knochentyp und Extraktionsmethode
Spalten: STR-Systeme
Ausschnitt; aus [1]
Y-Chromosomale STR-Systeme:
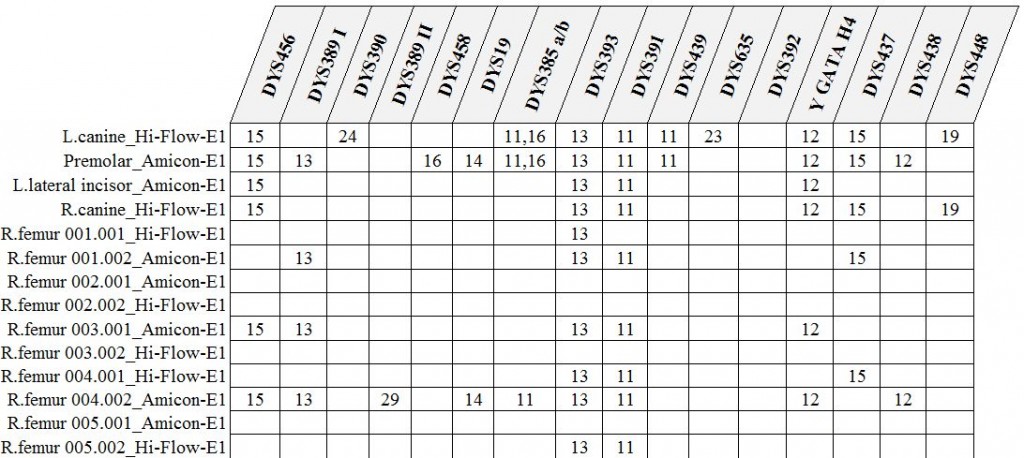
Zeilen: einzelne Proben, jeweils mit Knochentyp und Extraktionsmethode
Spalten: STR-Systeme
Ausschnitt; aus [1]
Die Tabellen zeigen jeweils Ausschnitte aus den autosomalen und y-chromosomalen STR-Ergebnissen. Man erkennt, daß für alle Knochen und die verschiedenen Extraktionsmethoden jeweils nur Teil-Profile erzeugt werden konnten, was auf die durch das hohe Alter der Knochen bedingte DNA-Degradation zurückzuführen ist. Es gelang aber, durch Zusammenführen der in allen Systemen jeweils übereinstimmenden Teilprofile, sogenannte Konsensus-Profile für autosomale und Y-chromosomale STR-Systeme zu erstellen, so daß für den Verstorbenen jeweils vollständige Profile rekonstruiert werden konnten. Die beste Extraktionsmethode schien übrigens die organische Extraktion gewesen zu sein, die ansonsten und eigentlich kaum noch Verwendung in der forensischen Routine findet. Der Grund ist wahrscheinlich, daß bei diesem Verfahren am wenigsten DNA verloren geht.
Da der mögliche Sohn von Zeke Harper, Earl, nicht exhumiert wurde, standen keine direkten Verwandten Zekes als Vergleichspersonen zur Verfügung. Von Earls sieben Kindern lebte nur noch eine seiner Töchter, die allerdings kein Y-Chromosom besitzt. Außerdem standen noch zwei mögliche Urenkelsöhne Zekes zur Verfügung, die jeweils von einem Sohn Earls abstammten und demnach Zekes Y-Chromosom über drei Generationen von ihm geerbt haben müssen.
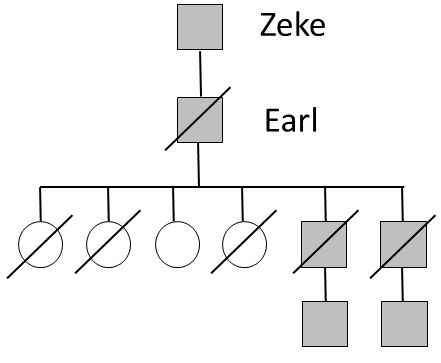
Quadrat: Mann, Kreis: Frau; durchgestrichen: steht nicht zum Test zur Verfügung; grau schattiert: gemeinsames Y-Chromosom
In einer solchen Konstellation bringt eine Einbeziehung autosomaler STR-Systeme kaum Nutzen, da bei diesen Systemen die Rekombination über zwei bzw. drei Generationen viel zu hoch ist, um auf überzeugende Werte für die Wahrscheinlichkeit einer möglichen Verwandtschaft zu kommen. Also konzentrierte man sich auf die Y-Chromosomen. Wie gesagt: wenn Earl wirklich der Sohn Zekes war, dann müssen Zeke und er und alle von ihm, Earl, abstammenden Männer, also auch seine Enkel, einen (bis auf mögliche Mutationen) identischen Y-chromosomalen Haplotypen besitzen.
Genau das wurde im vorliegenden Fall gefunden: der Y-chromosomale Haplotyp der beiden Enkel Earls stimmte untereinander und mit dem rekonstruierten Y-chromosomalen Haplotypen des verstorbenen Zeke vollständig überein! Nun mußte noch überprüft werden, mit welcher Wahrscheinlichkeit diese Übereinstimmung zufällig zustande kam, was davon abhängig ist, wie häufig genau dieser Y-chromosomale Haplotyp in der Population, aus der Zeke und seine Nachkommen stammen, ist. Dazu konsultierten die Autoren zwei unabhängige Datenbanken für Y-Haplotyp-Häufigkeiten (USYSTR und YHRD). In keiner der Datenbanken wurde der Haplotyp gefunden. Aufgrund der Größe der Datenbanken läßt sich daraus berechnen, wie selten der Haplotyp in der Population ist. Demnach ist es z.B. bei einer zugrunde gelegten europäischen Metapopulation 3333 mal wahrscheinlicher, diesen Y-Haplotypen in der Knochen-DNA und den beiden möglichen Urenkeln zu finden, wenn Zeke und Earl in väterlicher Linie verwandt sind, als wenn sie es nicht sind.
War also Zeke nun Earls Vater? Die vorsichtige Antwort lautet: die Evidenz ist mit dieser Hypothese vereinbar 🙂
_____
Referenzen:
[1] Ambers, A., Gill-King, H., Dirkmaat, D., Benjamin, R., King, J., & Budowle, B. (2014). Autosomal and Y-STR analysis of degraded DNA from the 120-year-old skeletal remains of Ezekiel Harper. Forensic Science International: Genetics, 9, 33-41.
[2] Loreille, O. M., Parr, R. L., McGregor, K. A., Fitzpatrick, C. M., Lyon, C., Yang, D. Y., … & Robinson, E. M. (2010). Integrated DNA and Fingerprint Analyses in the Identification of 60‐Year‐Old Mummified Human Remains Discovered in an Alaskan Glacier. Journal of forensic sciences, 55(3), 813-818.






Kommentare (4)