Zweieinhalb Jahre ist es her, daß ich ein interessantes Gespräch mit Rudolf Jaenisch führte, anläßlich der Verleihung der Otto-Warburg-Medaille an ihn. Jaenisch ist ein Molekularbiologe, dessen wissenschaftliche Schwerpunkte u.a. die Erforschung der epigenetischen Mechanismen verschiedener Krankheiten, darunter Krebs, umfassen. (Hinweis: um den folgenden Artikel besser zu verstehen, sind Kenntnisse der Konzepte von CRISPR/Cas und Epigenetik sehr hilfreich.)
Im Rahmen unseres Gesprächs sprachen wir auch über das revolutionäre System zur Genomeditierung, genannt CRISPR/Cas, das er verwendet hatte, um iPSCs zu erzeugen und schon damals als „game changer“ bezeichnete.
Seit unserem Gespräch waren Jaenisch und seine Gruppe offenbar nicht untätig: kürzlich erschien in der Zeitschrift Cell ein Artikel darüber, wie man CRISPR/Cas nutzen kann, um DNA-Methylierungsmuster zu verändern [1].
Seit langem ist bekannt, daß methylierte Bereiche in der DNA aber auch auf DNA-bindenden Proteinen wie Histonen die Genexpression beeinflussen (gewöhnlich reduzieren, manchmal aber auch aktivieren) können. Im Rahmen großer internationaler Projekte wie ENCODE und dem „Roadmap Epigenomics Project“ gelang es, die DNA-Methylierungsmuster in hunderten von Zellinien und primären Zellen zu katalogisieren, wodurch das Verständnis dessen, wie durch Methylierungen die Genexpression reguliert wird, vertieft wurde. Was aber zur Vervollständigung des Bildes fehlte, war das Verständnis der funktionalen Bedeutung einzelner Methylierungsereignisse. Um diese zu begreifen, müßte man in der Lage sein, ganz gezielt die DNA-Methylierung zu verändern.
Und genau das ist Jaenischs Gruppe nun gelungen. Sie berichten in ihrem Artikel [1], daß sie das CRISPR/Cas-System umfunktioniert haben, um Methylgruppen in die DNA einzufügen oder zu entfernen, so daß die Funktion einzelner Methylierungen, z.B. in der Entwicklung oder ihre Rolle bei bestimmten Krankheiten untersucht werden kann. Dies würde zugleich eine Möglichkeit zur Behandlung epigenetischer Erkankungen erschließen.
Jaenisch und Co. sind übrigens nicht die Einzigen, die auf diese Idee gekommen sind. Andere Gruppen haben modifizierte CRISPR/Cas-Systeme benutzt, um die Expression von Genen des Immunsystems, für Knochenumbau und im Zusammenhang mit Brustkrebs zu regulieren [2-4].
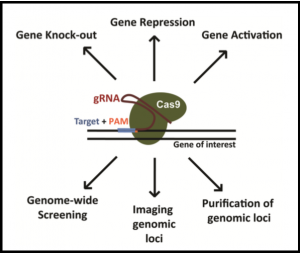
Die Abbildung deutet die Vielseitigkeit des CRISPR/Cas-Systems an, die durch verschiedene Modifikationen von Cas9 entsteht und neben Gen-Knockout, -Repression und -Aktivierung auch genomweites Screening, die Darstellung und Reinigung einzelner Loci gestattet. Und eben auch die Methylierungsmodifikation von DNA (c) cdn2.hubspot.net
Alle diese Ansätze beruhen auf einer Modifikation der Nuklease Cas, die als „tot“ (engl. dead) bezeichnet und dCas abgekürzt wird. Diese Cas-variante ist katalytisch „tot“, schneidet also die DNA nicht mehr, sondern agiert wie eine Plattform für andere Proteine, nachdem sie durch die gRNA an die richtige Stelle dirigiert wurde. Die dCas-Variante ist schon länger bekannt und wurde u.a. benutzt, um die Transkription zu steuern und sogar, um DNA in bestimmten Bereichen anzufärben. Für die Methylierungsmodifikation wird entweder eine bestimmte DNA-Methyltransferase (DNMT3a) an dCas angefügt, die dann Methylgruppen auf die DNA übertragen kann, was in den meisten Fällen die Stillegung eines Gens zur Folge hat. Oder es wird eine bestimmte Methylcytosin-Dioxygenase (TET1) an dCas angefügt, die in der Lage ist, Methylgruppen von Cytosinbausteinen in der DNA zu entfernen, wodurch in den meisten Fällen ein stillgelegtes Gen wieder aktiviert wird.
Die Gruppe um R. Jaenisch hat diese Werkzeuge nun genutzt, um Methylgruppen aus der Promotorregion des „brain-derived neurtrophic factor“-Gens (BDNF), das eine Rolle für Lernen und Gedächtnisbildung spielt, in Mäuseneuronen zu entfernen, wodurch das Gen aktiviert wurde. In einem anderen Experiment benutzten sie das dCas-System, um ein künstlich in eine Mauszellinie eingebautes, durch Methylierung reguliertes Reportergen zu aktivieren. Das Gen besteht aus einem Promotor mit „Methylierungsschalter“ und einem kodierenden Teil für ein fluoreszierendes Protein (die leuchten, wenn sie mit einer bestimmten Lichtwellenlänge aktiviert werden, ein besonders bekanntes Beispiel ist GFP). Dieses Reportergen kann an einer beliebigen Stelle im Genom eingebaut werden und sein Methylierungsstatus kann dann bis auf Einzelzellebene aufgelöst werden, indem das Leuchten des fluoreszierenden Proteins unter dem Mikroskop beobachtet wird [5].
Mit dem System können so die Methylierungsmuster in ganz verschiedenen Zelltypen modifiziert werden und die nächsten Schritte werden sein, das System an einem lebenden Tier zu testen. So ließe sich vielleicht untersuchen, ob bestimmte Methylierungsmuster an der Kodierung oder Speicherung von Gedächtnisinhalten beteiligt sind.
______
Referenzen:
[1] Liu, X. S., Wu, H., Ji, X., Stelzer, Y., Wu, X., Czauderna, S., … & Jaenisch, R. (2016). Editing DNA Methylation in the Mammalian Genome. Cell, 167(1), 233-247.
[2] Vojta, A., Dobrinić, P., Tadić, V., Bočkor, L., Korać, P., Julg, B., … & Zoldoš, V. (2016). Repurposing the CRISPR-Cas9 system for targeted DNA methylation. Nucleic acids research, gkw159.
[3] Xu, X., Tao, Y., Gao, X., Zhang, L., Li, X., Zou, W., … & Hu, R. (2016). A CRISPR-based approach for targeted DNA demethylation. Cell Discovery, 2.
[4] Choudhury, S. R., Cui, Y., Lubecka, K., Stefanska, B., & Irudayaraj, J. (2016). CRISPR-dCas9 mediated TET1 targeting for selective DNA demethylation at BRCA1 promoter. Oncotarget, 7(29), 46545-46556.
[5] Stelzer, Y., Shivalila, C. S., Soldner, F., Markoulaki, S., & Jaenisch, R. (2015). Tracing dynamic changes of DNA methylation at single-cell resolution. Cell, 163(1), 218-229.



Kommentare (8)