Meine Lieblingsmathematik-Kolumne gibt es in der niederländischen Zeitung “Volkskrant“. Und zwar “Ionica zag een getal“, also “Ionica sah eine Zahl” von Ionica Smeets. Witzig und abwechslungsreich, Rätsel, ernsthafte Mathematik, Sonstiges, für eine Tageszeitung ungewöhnlich – mir gefällt ihr Stil. Vielleicht beim nächsten Urlaub in “Holland” bzw. NL am Kiosk mal einen Blick riskieren?

Professorin für Wissenschaftskommunikation Smeets (so was gibt es!) aufgenommen von Ype Driessen — Abbildung erlaubt, sofern der Fotograph genannt wird.
Letztens gab es eine Kolumne, deren Titel meine Aufmerksamkeit erregte: Es ist eine Beschwerde, dass Antragsschreiben so viel Zeit und Mühe kostet.
Das ist ein Lamento, so alt wie das Antragsschreiben selbst. Immer wieder gehört. Wer wird ihr nicht recht geben? Ich jedenfalls sehe das ganz ähnlich: Angesichts der Prophetie, die allen Anträgen innewohnt, wird immer mehr hinterfragt, ob der status quo bei Antragsverfahren noch sinnvoll ist. Sogar über eine Lotterie wird nachgedacht: Denn, warum nicht Lotterie nennen, was Lotterie ist?
Mich treibt noch eine andere Sache um: Bei verschiedenen „hallway tracks“ habe ich auch die oder den GruppenleiterIn auf den miesen Stand der Software in der Bioinformatik angesprochen. In der Bioinformatik, der akademischen Softwareentwicklung allgemein, wird oft proof-of-concept-Software geschrieben und veröffentlicht: Man hat gezeigt, dass man ein bestimmtes Problem lösen kann. Diese Lösung wird veröffentlicht, dann gibt es das nächste Projekt – eine Konsequenz des publish or perish, die auch andere Felder heimsucht.
Lennart Martens hat die Problematik in einer Präsentation sehr schön illustriert:
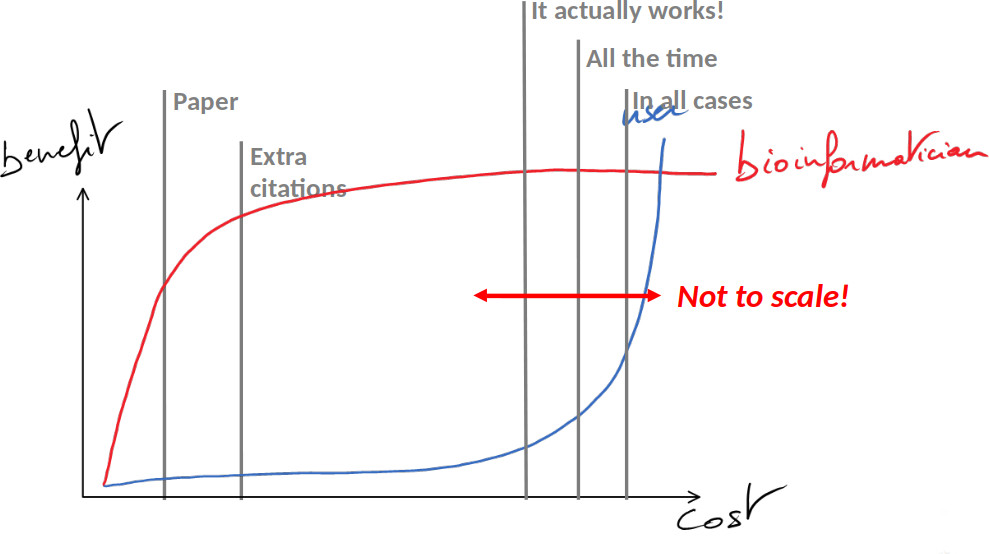
Skizze von Lennart Martens (frei unter CC-BY 4.0; Danke Lennart!): Aus Sicht der wissenschaftlich arbeitenden (Bio-)InformatikerInnen zählt das Paper zu einer Software, vielleicht noch das Aufbessern mit zusätzlichen “Features”, damit auch Zitate kommen. Alle weitere Arbeit bringt wenig. NutzerInnen hingegen interessiert: Funktioniert das Programm? Immer? Wie komme ich damit (schnell) zu meinem Paper?
AnwenderInnen wissenschaftlicher Software brauchen verlässliche Software. Fehler gibt es immer wieder, da ist Korrektur und somit jemand die oder der die Fehler „fixt“ unabdingbar für den Erfolg der Software (also die lange Nutzung durch viele). Doch wie viele Softwareprojekte sind schon bei Erscheinen Ihrer Veröffentlichung verwaist, werden also nie gewartet? (Ich bin bei einer Stichprobe in meinem Sektor auf eine Zahl >> 50 % gekommen – eine systematische Erhebung ist in Arbeit.)
Die DFG hat den Bedarf erkannt. Das ist schön. Ob das an der Gesamtsituation hierzulande etwas ändert? So lange die Entwicklung wissenschaftlicher Software fast nicht gefördert wird, wird auch oft von Doktoranden ohne IT-Ausbildung “nebenher” dran gearbeitet. Gute Software, Software, die schnell, parallel und verlässlich arbeitet, kann so selten entwickelt werden. Die DFG führt zurzeit die Wahl ihrer Fachkollegien durch. Ich kenne dort nicht eine Kandidatin, einen Kandidaten der / dem selber an nachhaltiger Softwareentwicklung für die Wissenschaft gelegen ist – was aber hoffentlich nur daran liegt, dass ich so wenige Kandidaten persönlich kenne. Über gegenteilige Infos, ggf. sogar Kontakte, würde ich mir sehr freuen!
Jedenfalls war man mal weiter, z. B. mit den „collaborative computation projects“ (CCP) aus UK. CCP4 lebt immer noch, also über 40 Jahre! Wow! Ok, ich weiß nicht welche Paradigmenwechsel während 40 Jahren in einem Projekt vollzogen wurden, geschweige denn wie die Personalstärke bzw. Finanzierung über diesen Zeitraum ausgesehen hat, dennoch: Hut ab! Und dann gibt es auch noch Molekulardynamik-/ab initio-simluationen. Da gibt es auch einige Beispiele für Langlebigkeit. Oder finite Elemente: Da auch (ich erhebe natürlich keinen Anspruch auf Vollständigkeit). Oder, oder, oder … Was diese erfolgreichen, gut gepflegten Projekte gemeinsam haben:
– es gibt nicht nur eine(n) EntwicklerIn
– es geht nicht um Bioinformatik
Ausnahmen bestätigen natürlich die Regel, doch gehorcht die (genomorientierte) Bioinformatik letztlich oft anderen Paradigmen (ihr Probleme sind meist nicht „eng gekoppelt“): Ihre Datenanalyse ist in der Regel a) mehrstufig und b) sind die Einzeldatensätze unabhängig voneinander.
Davon bald mehr – bis dahin: Hoffentlich nehmen diejenigen, deren wissenschaftliche Arbeit unter schlechter Software leidet, den Ball in den Gremien auf und lenken die Vergabe der Mittel entsprechend – damit die Zeit beim nächsten Schreiben ihrer Anträge wenigstens gut investiert ist.



Kommentare (3)