Gerade lernen wir, dass Covid-19 wohl auch erhebliche Folgeschäden provozieren kann. Beispielsweise kann das SARS-CoV-2-Virus die Nieren stark schädigen [Puelles et al., 2020], mögliche Lähmungen des Atemsystems verursachen und macht vor der Psyche nicht halt[Rogers et al., 2020]. Vor allem aber kann es auch starke Schädigungen der Lungen geben[Ye et al., 2020] – was wir schon recht früh wussten. Grund genug also an allen Fronten zu forschen und die Pandemie einzudämmen. Aktiv gegen das Virus werden können wir – das ist bekannt – auf drei Ebenen:
-
- Impfstoffe erforschen, auch wenn dies möglicherweise wenig aussichtsreich ist.
- Die Behandlung der Patienten optimieren. Mittlerweile (Mai 2020) scheinen Tests auf die Behandlung mit Gerinnungshemmern en vogue zu sein, wofür es gute Gründe gibt[Wichmann et al., 2020].
- Neue Wirkstoffe erforschen bzw. neue Anwendungen für bereits etablierte Medikamente finden.
Auf der gesellschaftlich aktiven, aber medizinisch passiven Seite stehen außerdem und wohlbekannt die sozialen und politischen Maßnahmen – heiß diskutiert.
Hier und heute soll es um den dritten Punkt gehen, hat der es doch bereits zu einem Wikipediaeintrag gebracht und einen berühmten, wenngleich idiotischen Befürworter im Kontext der Covid-19-Prophylaxe. Unter dem Stichwort Umwidmung – drug repurposing – wird nicht erst seit “Corona” mit der Suche nach neuen Anwendungen für etablierte Medikamente versucht Therapienotstände zu mildern, gar nicht erst entstehen zu lassen oder überhaupt ein Medikament für seltenere Krankheiten zu finden. Insbesondere auch, weil auf diese Weise viel Geld gespart werden kann – schließlich sind Entwicklung, Darreichungsformen geklärt und auch Phase I-Studie schon durchlaufen – teils kann auch die Phase II “recycelt” werden.
Solch profanen Überlegungen sind aktuell – für viele von uns – zweitrangig. Die wissenschaftliche Community hat die Notwendigkeit verstanden (und die Gelegenheit vielzitierte Veröffentlichungen zu produzieren) und forscht mit Hochdruck an Lösungen rund um die aktuelle Pandemie – von Soziologie bis zur Therapieforschung sind alle dabei.
Bei der Entwicklung antiviraler Wirkstoffe steht die Frage im Vordergrund “An welche Bestandteile – des Virus oder der Wirtzellen – können Wirkstoffe überhaupt binden – und wenn sie dies tun, mit welchem wahrscheinlichen Effekt?” Kollegen aus Frankfurt sind dieser Frage mit proteomischen Mitteln nähergekommen[Bojkova et al., 2020]. Sie haben untersucht welchen Weg das Virus in der Wirtszelle beschreitet – auf der Proteomebene. Was das heißt? Die Gruppen haben Zellkulturen (also menschliche Zellen in der Petrischale) mit dem Virus behandelt und dann mittels sogenannter “mePROD”-Technik untersucht, welche Proteineproduktion nach der Infektion der Zellen runter- oder hochgeregelt wird. Bei diesem Vorgehen trennt man die Zellen in bestimmten Zeitabständen auf, trennt deren Bestandteile chromatographisch, also mit einer flüssigkeitsgefüllten Säule, auf und bestimmt so welche Bestandteile viel oder wenig vorkommen. Damit anschließend erkannt werden kann, was genau diese Bestandteile sind, werden diese mittels Massenspektrometrie analysiert, denn Bruchstücke von Eiweißen können anhand ihres Gewichtes gut den Eiweißen (Proteinen) zugeordnet werden. So konnte gezeigt werden, dass Wirkstoffe, die die sog. Translation (also die Übersetzungsmaschienerie für den Zusammenbau von Eiweißstoffen) hemmen, die Virusproduktion der Zellen stark stören, die Zellen aber nicht umbringen. Außerdem hat die Gruppe bestätigt, dass die Menge des ACE2-Rezeptor nach einer Infektion geringer wird, weil der zerstückelt wird (hatte ich vormalig in einer Spekulation bereits erwähnt) – den braucht das Virus um an seine Wirtszellen (also unsere Zellen) zu binden.
Wichtig für uns ist: Einer der möglichen Hemmstoffe ist Ribavirin – und das wird bereits in einer Studie untersucht. Das muss möglicherweise nicht viel bringen, denn wir erinnern uns das Remdisivir auch mit großem Getöse als möglicher Heilsbringer gegen COVID-19 angekündigt wurde, um anschließend mit eher mauen Ergebnissen in der Presse zu stehen (derselbe Link zeigt auch, dass noch etwas Warten angebracht ist, um ein abschließendes Urteil zu fällen). Also gilt es weiter zu suchen:
Diese antiviralen Wirkstoffe wurden auch bereits mit molekularen Docking untersucht[Elfiky 2020]. Hierbei handelt es sich um eine informatische Methode. Das “Experiment” wird im Computer durchgeführt. Üblicherweise dient es dazu unter tausenden oder gar viele Millionen möglicher Wirkstoffsubstanzen diejenigen zu finden, an denen es lohnend sein könnte weiter zu forschen. Diese Einschränkung ist wichtig, denn was untersucht wird, ist Folgendes: Für jedes der vielen Docking Versuche werden im Computer zwei Moleküle zusammengebracht. Häufig (und auch hier) ein Protein (groß) und ein kleines Molekül (dem sog. Liganden). Im Idealfall ist die Ausgabe eine Stellung (Konformation) in welcher der Ligand bindet und die bei der Bindung freiwerdende Energie (die sog. Bindungsenthalpie). Und weil die beteiligten Moleküle meist komplex sind, kann man das nicht direkt berechnen, sondert verwendet ein Näherungsverfahren. Einzelne Docking-Versuche können mit ein bisschen Geduld am Desktop-PC durchgeführt werden. Bei sehr großen Zahlen von Liganden und einigen möglichen Proteinpartnern allerdings braucht es große Computer, Supercomputer, und weil so viele Substanzen beteiligt sein können, spricht man auch von screening, also von einem Sichten der Substanzen.
Meine Kollegen Onat Kadioglu und Mohamed Saeed aus der Arbeitsgruppe von Professor Efferth in Mainz haben genau das getan, zusammen mit Henry Johannes Greten aus Heidelberg[Kadioglu et al., 2020]. Also viele mögliche Liganden und manche Proteine des SARS-CoV-2-Virus auf ihre wechselseitige Bindung hin untersucht. Das ging auch bereits durch die Presse (Beispiel). Der wesentliche Unterschied zur Veröffentlichung von Elfiky ist, dass mehr Kombinationen abgesucht wurden. Zum Einen wurden die SARS-CoV-2 spezifischen Proteine modelliert (weil es noch keine Strukturen der spezifischen SARS-CoV-2-Varianten gibt): Das ‘spike‘ Protein (was die Corona, des “Coronavirus” ausmacht), Das Nucleokapsid (welches mit der RNA des Virus assoziiert ist) und die 2’-o-Ribose Methyltransferase (ein Enzym, das die RNA des Virus bearbeitet). Wichtige Proteine also, denn ohne die Funktion dieser Proteine, kann sich das Virus nicht vervielfältigen (lassen).
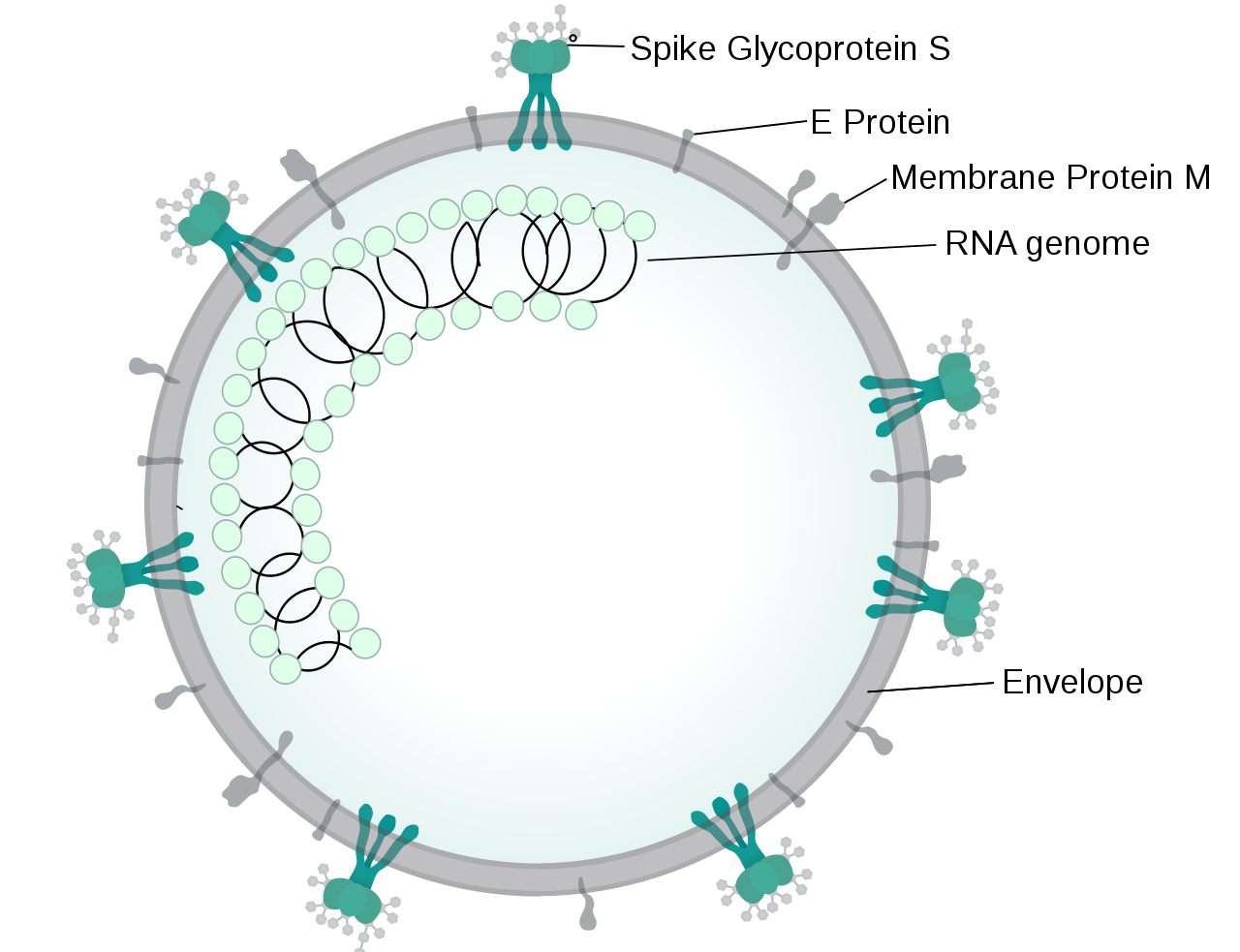
Morphologie des SARS-CoV-2-Virus (Quelle: Wikipedia, Lizenz CC BY-SA 4.0).
Vor allem wurden etwas mehr (noch nicht richtig viele) Substanzen untersucht – nur damit ihr eine Vorstellung davon habt, was “nicht viel” hier bedeutet: 1577 bereits von der FDA zugelassene Stoffe, 39442 Naturstoffe aus der ZINC-Datenbank und 115 weitere Naturstoffe. Insgesamt also Kombinationen.
Ein weiterer Unterschied dieser Arbeit zu der Arbeit von Elfiky besteht vor allem darin, dass Negativkontrollen mit berücksichtigt wurden: Wenn man recht sicher weiß, dass ein Stoff sich nicht als Wirkstoff eignet kann dieser dennoch an das Zielprotein binden und so können das auch andere Stoffe; teils unspezfisch. Durch den Ausschluss der Bindung von guten Negativkontrollen ist man sich also sicherer als ohne, dass die gefundenen Kandidatenstoffe Potential als antivirale Wirkstoffe haben.
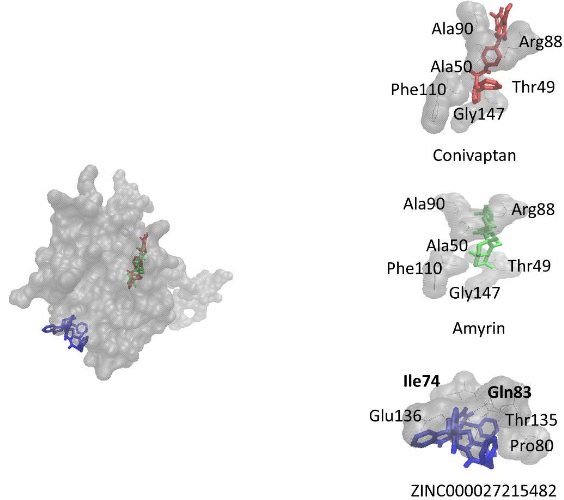
Und was wurde gefunden? Einige Substanzen, die im anderen Kontext wirken (z. B. als Krebstherapeutika), aber auch vor allem Anti-Hepatitis-C-Medikamente, die als potentielle Wirkstoffe gegen den verwandten MERS-Virus wirksam sind. Ein Resultat ist in der Einganggraphik zu sehen: Ihr seht hier drei verschiedene Stoffe und ihre Bindung an das Nucleokapsidprotein. Wer mehr wissen will, das Original (und die anderen Veröffentlichungen) möchte ich wirklich ans Herz legen – es ist gut geschrieben und in diesem Blogbeitrag hoffte ich die “Verständnislatte” nicht zu hoch zu hängen.
Was ich jetzt gerne tun würde, ist die erforderlichen Skripte zusammen mit den Kollegen in einen gescheiten HPC-konformen Workflow giessen, denn zukünftige Projekte (es gibt ja noch mehr zu erforschen als mögliche antivirale Stoffe und auch da ist noch Potential für mehr) würden von verbesserter Nutzbarkeit stark profitieren. Die Arbeit (den Workflow mitzuentwickeln) ist bereits ausgeschrieben …



Kommentare (7)