Zwei Nobelpreise hat sie schon gebracht, die Elektronenmikrospie. Und doch hat sich in den letzten Jahren eine unglaubliche Revolution, die sog. resolution revolution oder Auflösungsrevolution vollzogen.
In meiner Diplomarbeit habe ich noch 2D-Elektronenkristallographie gemacht und war froh eine Projektionskarte eines Natrium-Protonen-Austauschproteins (s. o.) von E. coli-Bakterium mit 6 Ångström (1/10 nm, meiner Lieblings-nicht-SI-Einheit mit der die Welt der Strukturbiologie heute noch arbeitet) zu erhalten. Elektronenkristallographie ist eine Methode zur Bestimmung der Struktur von Membranproteinen; Proteinen also, die ihre Funktions in Zellmembranen ausüben. Einige Jahre zuvor hatte dieselbe Arbeitsgruppe diese bereits veröffentlich[Williams et al., 1999].Damals gab es für diese Art von Projektionskarten noch relativ hochrangige Zeitschriften, wo diese Art von Arbeiten veröffentlicht werden konnten:
Die Kristallisation von Membranproteinen ist eine aufwendige Sache und so ist eine Platzierung eines Artikels in einer guten Zeitschrift berechtigt gewesen. Doch weil die Einsicht in die Funktionsweise eines Membranproteins bei einer Projektskarte dieser Auflösung doch recht eingeschränkt ist nenne ich die Zeitschrift auch für damalige Verhältnisse “hochrangig”. Man sieht lediglich, wie Untereinheiten zueinander stehen und kann eventuell abschätzen wie viele gewendelte Strukturen, sogenannte Helices durch die Membran gehen. Dürftiges Wissen, dass man auch mit biochemischen Methoden erlangen kann.
Später dann gab es zu diesem Protein eine 3D-Struktur, ebenfalls durch Elektronenkristallographie bestimmt und sogar in der Zeitschrift Nature veröffentlicht[Williams, 2000]. Und doch habe ich als Student erneut daran versucht. Warum? Weil die Auflösung zu gering war, um Aussagen über die Funktionsweise zu treffen. Und so habe ich (erfolglos) versucht mit biochemischen Markierungen zu erfahren wo in den Elektronendichteprojektionen welche Position der Sequenz zu finden ist. Viel Aufwand für nichts, denn noch später konnte eine hochauflösende 3D-Struktur gewonnen werden[Appel et al., 2009].
Die Revolution in der Einzelpartikelanalyse
Lange Zeit war Einzelpartikelartikelanalyse – bei der viele tausend einzelne Teilchen aus den Aufnahmen im Computer zusammengebaut werden zu dreidimensionalen Strukturen, mit Auflösungen bei denen man vielleicht sehen konnte welche Untereinheiten wie zueinander standen state of the art. Und doch war bei ~20-10 Å Schluss (je nach Symmetrie des untersuchten Moleküls und anderer Faktoren). Mittlerweile gelingt es Strukturen großer Proteinkomplexe bei atomarer Auflösung darzustellen – und das ist verdammt interessant, denn so kann man beispielsweise erkennen wie so ein Eiweißkomplex, der aus verschiedenen Bausteinen zusammengesetzt ist, reguliert werden kann (z. B. um Medikamente zu entwickeln). Doch wie es das möglich geworden?
Da ist zunächst einmal die Präparationsmethode, bei der die Moleküle in einem dünnen Eisfilm ins Mikroskop kommen. 1984 eine kleine Revolution[Adrian et al., 1984] – und ein Grund für den zweiten Nobelpreis. Dann aber mussten neue Algorithmen entwickelt werden und die Hardware verbessert werden. Erst mit mehr Rechenpower und verbesserter Aufnahmetechnik konnte es gelingen die Auflösung wirklich extrem zu erhöhen von ~20-25 Å bis auf unter 2 Å zu bringen[Yip et al., 2020].
Und doch ist die Elektronmikroskopie, wenngleich unglaublich stark bei der Analyse großer Proteinkomplexe, weiterhin eine Methode unter mehreren der Strukturbiologie. Insbesondere die Röntgenkristallographie wird weiterhin eine große Nische besetze: Verlässlich hohe Auflösungen bei kleinen und großen Proteinen garantieren dafür, auch wenn die Kristallisation immer wieder eine zu überwindende Hürde darstellt.
Elektronenmikroskopie und SARS-CoV-2?
Und heute? Heute bricht eine Pandemie aus und viele Arbeitsgruppen rund um die Welt bearbeiten Teilprobleme. Selbstverständlich auch die Strukturbiologen. Sehr schnell wurden Strukturen veröffentlicht, z. B. diese hier:
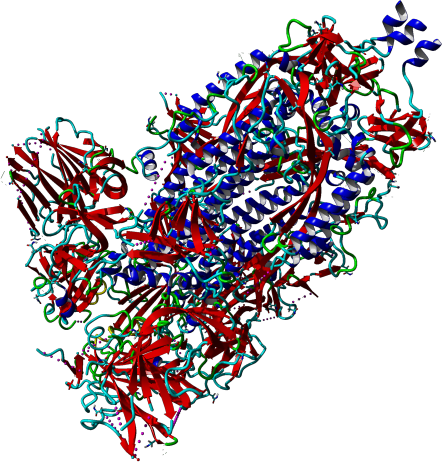
Bereits Ende Februar wurde diese Struktur des Spikeproteins in der sogenannten “geschlossenenen Form” (bzw. Konformation) veröffentlicht. Das ist wahnsinnig schnell: Ausgehend von der veröffentlichten Sequenz lief die molekularbiologische Vorbereitung an. Unmittelbar danach erfolgt Reinigung und elektronenmikroskopische Untersuchung. Und all dies in wenigen Wochen, wo zu meiner Zeit® für manche Schritte gerne mal ein Jahr benötigt wurde! (Selber geplottete Struktur nach dem PDB-Eintrag 6VXX – nach Walls et al., 2020)
Die Datenbank der Strukturbiologie hat eine schöne Seite mit COVID-19/SARS-CoV-2-Ressourcen. Unter anderem auch eine Seite mit den Strukturen – des Spikeproteins in verschiedenen Zuständen, mit und ohne Bindung zu Antikörperfragmenten, mit und ohne Bindung zu ACE2-Teilen und andere Eiweißstoffe des Virus. Vor allem fällt auf, dass viele Strukturen von Gruppen der Elektronenmikrospie beigetragen wurden. Das war vor einigen Jahren noch absolut undenkbar: So viele Strukturen, die es erlauben atomare Modelle zu bauen aus Einzelpartikelanalyse! Die hier im Beitrag gezeigte Struktur hat eine Auflösung von “nur” 2.8 Å, und die gepunkteten Linien zeigen Lücken im Modell. Das bedeutet für Medikamentenenwickler zusätzliche Arbeit (Lücken modellieren und ggf. Simulationen zur Energieminimierung) und kommt daher, dass relative flexibele Bereiche in den Molekülen einfach nicht gut erfasst werden können – ein Problem, dass Kristallographen seltener haben, da dort diese Bereiche häufiger im Kristall fixiert werden.
Dennoch zeigt die aktuelle Entwicklung wie die Elektronenmikrospie ihre Nische vergrößern konnte und mittlerweile hochkompetetiv ist. Eine rasante und spannende Entwicklung.



Kommentare (3)