Vor einer Weile habe ich ja bereits auf dem Journal BMC Bioinformatics herumgehackt. Jetzt wollte ich es genauer wissen und habe gleich drei Journals unter die Lupe genommen:
- Bioinformatics
- PLOS Compuational Biology
- und zum Vergleich nochmals BMC Bioinformatics
Eigentlich wollte ich mal einen wirklich systematischen Vergleich erreichen, vielleicht auch im Rahmen einer studentischen Arbeit – Metawissenschaft gewissermaßen – aber dazu kam es bislang noch nicht und viel Zeit hatte ich zwischen den Jahren auch nicht, darum habe ich nur jeweils 10 Arbeiten angeschaut, die versprachen eine neue Software zu veröffentlichen (s. unten). Das Ergebnis kann man zusammenfassend so darstellen:
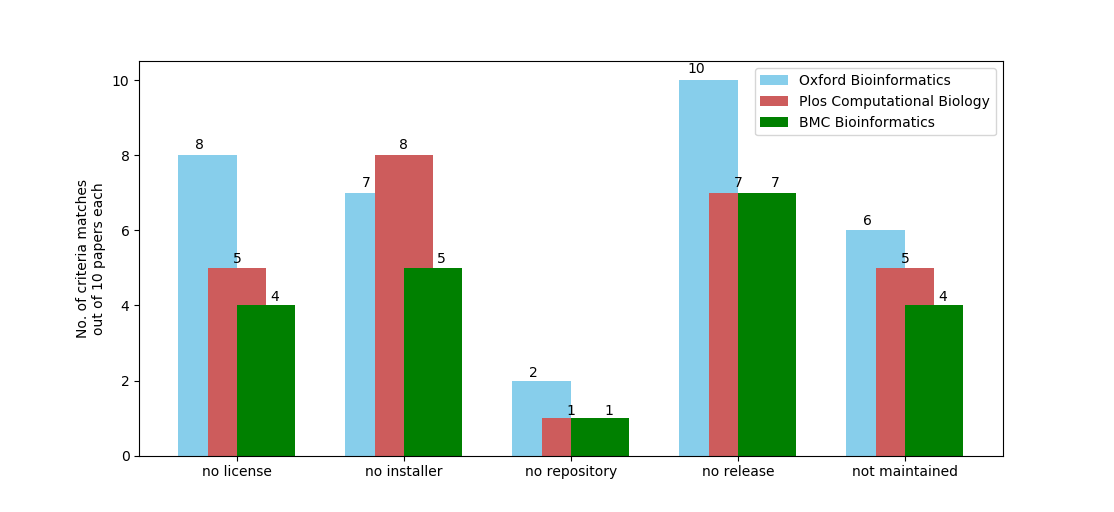
Hierbei wurde:
- eine Lizenz als fehlend gewertet, wenn es kein entsprechendes LICENSE oder COPYRIGHT File gab (ich habe nicht nachgeschaut, ob irgendwo ein entsprechender Text eingebettet war)
- eine Installationsroutine als fehlend gewertet, wenn es kein Makefile, CMakeLists-File, keine Python setup-Routine etc. gab. Bereitgestellte Container wurden nicht als Installationsroutinen gewertet. Ebenso wenig ein wie auch immer geartetes “ladet meinen Kram von github runter und startet es so wie es ist”.
- eine Softwarequelle (no repository) wurde als fehlend gewertet, wenn es keine gab (kein Link auf github, gitlab, etc. in der jeweiligen Veröffentlichung).
- eine Softwareversion/-veröffentlichung (no release) wurde als fehlend gewertet, wenn es kein versioniertes Paket gab.
- und schließlich wurde eine Software als nicht gewartet gewertet, wenn die letzten Änderungen zum Zeitpunkt des Nachschauens mehr als ein halbes Jahr zurück lag.
Erwartungen und Enttäuschungen
Seltsam, dass AutorInnen ohne Lizenz veröffentlichen, sollte man doch meinen ihnen ist daran gelegen, dass Dritte ihre Software auch nutzen. Darf man bloß ohne Lizenz eigentlich nicht. Und kann man auch nicht, wenn kein Installer da ist – zumindest nicht auf jeder Maschine. Auf einem Feld-Wald-Wiesen-Server einer Gruppe auf der beliebig viel Gammelcode sein darf, kann man ALLES installieren. Auf betreuten Multiusersystemen allerdings geht das nicht, da wird kontrolliert installiert – so dass auch Abhängigkeiten aufgelöst werden können und man sich auf bestimmte Versionen berufen kann (um es kurz auszudrücken).
Man sollte auch meinen, dass die Journals diese Sicht teilen, wird eine Arbeit doch kaum zitiert werden, wenn eine Software zu wenige Nutzer findet. Und in der Tat listen die Journals durchaus Bedingungen für Autoren, die eine Publikation erfüllen muss, so z. B. Bioinformatics:
Software or data must be freely available to non-commercial users
Jaaa, das geht ohne Lizenz? Oder
If describing software, the software should run under nearly all conditions on a wide range of machines.
Finde ich prima! Gibt es für dies und mehr denn eine Checkliste, eine Handreiche für Reviewer?
Ich habe nachgefragt und werde berichten.
Ein paar Schmankerl
Platz Nr. 3 meiner diesmaligen Charts geht an dieses Repository. Eine hübsch bebilderte Anleitung für Installation und Nutzung unter Windows – mit der ersten Verpflichtung eine Virtualbox von Oracle zu installieren. Und sehr eigene Vorstellungen welche Pfade zu nutzen sind.
Platz Nr. 2 geht an die Veröffentlichung in der steht
The raw data sets are large, and due to a hard drive crash many of the raw AutoDock output files were lost. However, the PDB used to generate the docking is provided as supplementary data. The AT1R PDB can be loaded to Model Archive (www.modelarchive.org) after acceptance of the manuscript.
Ist das nicht süß? Die Daten sind futsch, aber vielleicht wollen die Autoren die Resultate den KollegInnen zur Verfügung stellen. Was hatten die Reviewer eigentlich im Tee?
Platz Nr. 1 geht zweifelsohne an diese Software. Wow, kein Quellcodemanagement, kein Installer, keine Lizenz – all diese Pimperlitzchen braucht es nicht. Moderne Code Monkeys laden zu Google hoch. Jawoll! Geneigte Nutzer müssen dort den Code etwas für sich anpassen (sonst können sie ihre Daten nicht analysieren) und haben garantiert keine Möglichkeit des Datenmanagements, dafür aber ist alles in der Cloud! Sogar die ganzen Anfängerfehler an deren schlechtem Beispiel sich das Unterrichten lernen lässt. Wahnsinn!
++++++++++
Hier sind noch die Veröffentlichungen, die ich mir dieses Mal angeschaut habe:
Zunächst Bioinformatics (Ausgabe: Oktober 2020, die ersten zehn Artikel, die Softwareveröffentlichungen enthielten):
- https://doi.org/10.1093/bioinformatics/btaa252
- https://doi.org/10.1093/bioinformatics/btaa595
- https://doi.org/10.1093/bioinformatics/btaa599
- https://doi.org/10.1093/bioinformatics/btaa607
- https://doi.org/10.1093/bioinformatics/btaa609
- https://doi.org/10.1093/bioinformatics/btaa603
- https://doi.org/10.1093/bioinformatics/btaa032
- https://doi.org/10.1093/bioinformatics/btaa596
- https://doi.org/10.1093/bioinformatics/btaa606
- https://doi.org/10.1093/bioinformatics/btaa606
Dann Plos Computational Biology (Ausgaben: November/Dezember 2020, die ersten zehn Artikel, die Softwareveröffentlichungen enthielten):
- https://doi.org/10.1371/journal.pcbi.1007575
- https://doi.org/10.1371/journal.pcbi.1008277
- https://doi.org/10.1371/journal.pcbi.1007719
- https://doi.org/10.1371/journal.pcbi.1008386
- https://doi.org/10.1371/journal.pcbi.1008422
- https://doi.org/10.1371/journal.pcbi.1008003
- https://doi.org/10.1371/journal.pcbi.1008240
- https://doi.org/10.1371/journal.pcbi.1008349
- https://doi.org/10.1371/journal.pcbi.1008249
- https://doi.org/10.1371/journal.pcbi.1008247
Und BMC Bioinformatics (die letzten zehn Artikel, die den Software-Tag enthielten, Aufgenommen Dezember 2020/Januar 2021):
- https://doi.org/10.1186/s12859-020-03888-6
- https://doi.org/10.1186/s12859-020-03895-7
- https://doi.org/10.1186/s12859-020-03887-7
- https://doi.org/10.1186/s12859-020-03900-z
- https://doi.org/10.1186/s12859-020-03883-x
- https://doi.org/10.1186/s12859-020-03922-7
- https://doi.org/10.1186/s12859-020-03920-9
- https://doi.org/10.1186/s12859-020-03917-4
- https://doi.org/10.1186/s12859-020-03819-5
- https://doi.org/10.1186/s12859-020-03910-x



Letzte Kommentare