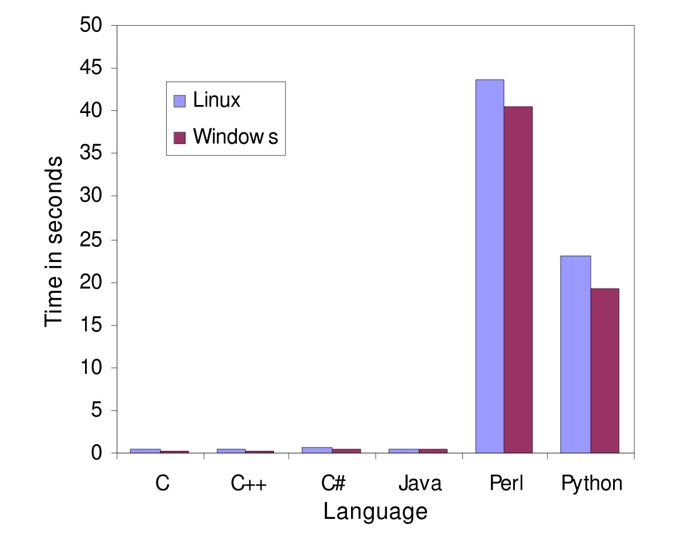
Geschwindigkeitsvergleich bei einem globalem Alignment; es werden also die Geschwindigkeiten bei einer Aufgabe verglichen, die in der Bioinformatik häufig vorkommt. Die Autoren geben an, stets gleichförmig implementiert zu haben. Lizenz: CC BY 4.0
Zwei Dinge werden unmittelbar deutlich:
- die beiden interpretierten Sprachen (Perl und Python) lösen das Problem deutlich langsamer als die anderen Sprachen.
- Linux scheidet konsequent schlechter ab.
Und weiter:
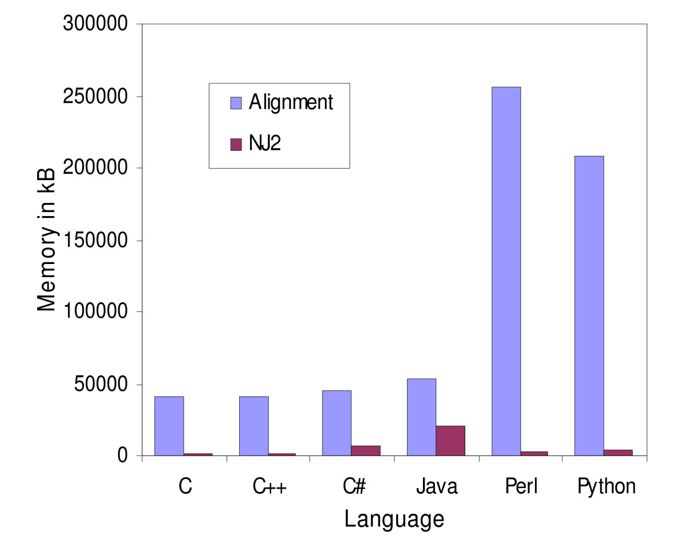
Die zweite Abbildung zeigt die Spitzen des Speicherverbrauchs beim gleichen Problem “Alignment” und bei einem bestimmten Gruppierungsverfahren. Lizenz: CC BY 4.0
Auch hier sieht man: Die interpretierten Sprachen benötigen deutlich mehr Speicher.
Über Benchmarks
Ein Disclaimer: Das Bonmot “Traue keiner Statistik, die Du nicht selber …” kennen wir alle. Bei Benchmarks, also dem systematischen Vergleich zwischen Programmen, Algorithmen, Implementierungen (also Umsetzungen in “Code”), usw. oder eben Programmiersprachen, verhält es sich ähnlich: Sie wecken schnell inneren Widerstand, weil schnell Gedanken aufkommen wie “die Leute haben X nicht berücksichtigt oder Y falsch gemacht”. Das liegt daran, dass eigene Erfahrungen und Wunschvorstellungen den Befunden häufig widersprechen. Womöglich ist der ein bestimmter Benchmark wirklich nicht gut. Aber man kann ja dennoch etwas daraus lernen.
Also weiter …
Der Vergleich “schummelt” ein wenig. Das Problem ist natürlich die Umsetzung: Wenn ich eine Aufgabe in Sprache X gut und in Y naiv umsetze, schneidet Y im Vergleich schlechter ab. Gute Programmiersprachenvergleiche setzen daher auf Streuung: Mehr ProgrammiererInnen, die sich jeweils eine Sprache, in der sie sich gut fühlen auswählen und damit Lösungen erstellen. Und solche Vergleiche sind deutlich differenzierter. Die Autoren obiger Veröffentlichung schreiben “All the programs examined here were written by the same programmer with different levels of experience in Java, Perl and C++. The other languages were implemented while learning them. …”. Da kann und sollte man Fragezeichen hinter manche Details setzen. Aber der allgemeine Trend — kompilierte Sprachen schnell, interpretierte langsamer und speicherhungrig — ist dadurch (für mich) nicht in Zweifel gezogen und ich finde das gerade auch nicht so wichtig: Mir geht es darum Unterschiede bei alltagsnahen Problemen aufzuzeigen – also alltagstauglich für programmierende BioinformatikerInnen. Und zu diskutieren.
Meine Erfahrung – leider (noch) nicht systematisch erhoben – ist:
- Java schneidet in puncto Speichernutzung oft richtig, richtig mies ab. Es gab in der Vergangenheit bei uns sogar Leute, die mehr als 80 GiB pro Rechnerkern (Core) veranschlagten – und das bei vielen, vielen Rechnungen**. Allerdings gibt es auch bessere Java-Programme in der Liga, die mit kompilierten Programmen mithält.
- PyPy, der just-in-time Compiler für Python, ist auch keine Lösung. Mag sein, dass Python so sehr viel schneller gemacht wird. Aber das Risiko die Geschwindigkeit mit erheblich höheren Speicherverbrauch zu erkaufen oder / und mit nicht portierbaren Lösungen dazustehen ist groß.
- Cython, ein Weg Pythoncode tatsächlich zu kompilieren und zu beschleunigen wird selten genutzt.
- Python wird oft derart mies umgesetzt, dass ich mich schon gedrängt gefühlt habe Hinweise zu geben, wie es besser geht – ohne, dass wir auf unserem Hochleistungsrechnern überhaupt Python als sog. Produktionscode (also Skripte, die den Kern von Berechnungen bilden und häufig laufen) sehen wollen – sie sind nicht nur Verschwendung teurer Resourcen, es ist auch unfair, wenn das langsamste Skript anderen Nutzern Rechenzeit nimmt. Um die Bioinformatik mal zu entlasten: Bei uns sind es auch theoretische PhysikerInnen, die schöne Algebra nutzen, diese 1:1 in Python gießen — und sich dann wundern warum die Skripte sooo langsam sind.
- Linux ist selbstverständlich nicht langsamer – das scheint mir eine Auswirkung der Compilerwahl und Einstellungen zu sein. Das Paper bleibt vage.
- Perl kommt in neueren Projekten nur noch selten vor. Es scheint an Bedeutung verloren zu haben.
- R fehlt in dem Vergleich völlig. Diese Sprache wird bei uns einfach so benutzt oder R-Pakete, wie Bioconductor, also eine Bibliothek, die viele Lösungen schon enthält. Bei R und insbesondere Bioconductor ist viel in C oder C++ implementiert und R dient nur als Schicht, die die AnwendungsprogrammiererInnen nutzen – mal kann man also sehr gute Performance erreichen, mal kann man Pech haben und eine naiv implementierte Bibliothek verwenden, die einen Flaschenhals mitbringt. Auch R bietet einen just-in-time Compiler, der bei uns allerdings selten genutzt wird.
Wieso ist das wichtig?
Meine Auswahl der Sprachen und Kenntnisse ist nicht willkürlich. Andere denken auch, dass diese Sprachen und zusätzlich Shellkenntnisse wichtig sind. Natürlich kann man über Details diskutieren. Und solchen Vergleichen wohnt immer der Funke zum Flamewar inne. Also, erst mal durchatmen …



Kommentare (50)