Wie ich schon vor ein paar Tagen schrieb: Ich unterrichte WissenschaftlerInnen aus nicht-IT-affinen Wissenschaften in Programmierung mit C++ und Python (und shell-Programmierung, etc.). Und das macht Spaß, ich hoffe sehr darauf, dass ich im Spätsommer oder Herbst die Kurse wieder in einem Kursraum mit lauter motivierten TeilnehmerInnen halten kann. Es wird eine große Erleichterung sein, bei den Übungen durch die Reihen zu gehen und unmittelbar Hilfestellung zu geben, wenn es irgendwo klemmt.
Und das es beim Programmieren klemmt, wird unweigerlich vorkommen. Wer irgendwann mal den Versuch unternahm das Programmieren zu lernen, kennt dies ebenso gut, wie professionelle ProgrammierInnen. Der Unterschied ist natürlich, dass Profis – gleich in welchem Bereich – eher wissen, wie man sich selber hilft oder Hilfe sucht. Deshalb kommen zu Anfang interaktive Oberflächen sehr zu pass: Wenn Anfängerinnen einen Befehl eingeben können (oder eine Funktion deklarieren) und dann gleich sehen, was dieses Bisschen Code tut (und ob das Erwartete herauskommt), dann ist dieser unmittelbare Feedback ein Riesenvorteil. Das Prinzip, was man hier nutzen kann, nennt man REPL – read eval print loop – und ist genau das, was ich gerade beschrieben habe. Hier ein Beispiel mit dem Python-Interpreter:
>>> 42*7*6 1764
Hierbei ist das ‘>>>’ der sogenannte Python-Prompt. Alles was nach einem “Prompt” kommt wird interpretiert. Hier haben wir den Python-Interpreter als Taschenrechner verwendet. Das Schöne ist, dass man sehr viel mehr interaktiv testen kann als nur dies Bisschen Grundschulmathematik: Alles was die Sprache bietet. Und dann kam vor ein paar Jahren Ipython auf und sehr früh auch das Iypthon-Notebook. Das Ipython-Notebook, später aufgegangen im Jupyter-Notebook (ab hier, kurz: “Notebook”), ist eine Browseranwendung: Ihr könnt darin auch Textfelder mit freien Kommentaren schreiben. Das ist einfach, aber ziemlich cool, wie wir gleich sehen werden. Das Notebook erlaubt es außerdem auch andere Sprachen, z. B. shell, R oder Julia, auszuführen – daher auch der Name “Jupyter”: Julia, Python und R.
Das Jupyter-Notebook in der Lehre
Viele LehrerInnen nutzten die Möglichkeiten des Notebooks. Neben einem Unterrichtseinsatz mit hohem interaktiven Anteil – man kann wunderbar in diese Notebooks Aufgabenstellungen und Vorbereitungen (Modulimporte, Beispieldaten) einflechten und vor Stundenbeginn verteilen – kann man natürlich auch Demonstrationen bieten und komplexe Sachverhalte visualisieren. Und das alles mit einem Werkzeug:
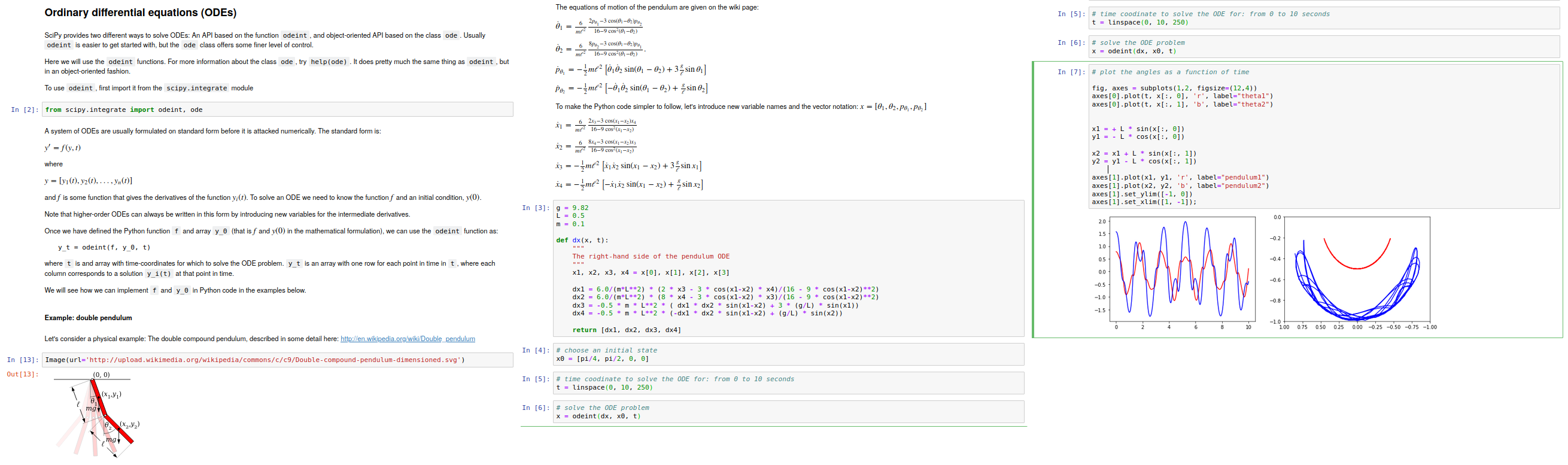
Am Ende meines Pythonkurses – der nicht wissenschaftliche Software-Bibliotheken zum Inhalt hat – gebe ich einen Ausblick auf die Möglichkeiten dieser Pakete: Lösen von Differentialgleichungen / einfaches Integrieren, Fouriertransformationen, spezielle Funktionen (wie z. B. Besselfunktionen) oder auch das Beispiel vom Doppelpendel, das hier gezeigt ist. Wie man sieht (drei Screenshots nebeneinander), erlaubt das Notebook auch mathematische Gleichungen mittels LaTeX sauber auszuschreiben und so einzuführen, wie man sie aus den Lehrbüchern kennt. Man darf es nicht übertreiben – auch programmierende WissenschaftlerInnen sind irgendwann müde und es fehlt die Motivation Gleichungen an der “Tafel” durchzukauen, aber hier geht es ja nur darum den Funktionsumfang des Jupyter Notebooks zu demonstrieren – soweit das in einem Blog überhaupt möglich ist. Man kann auch von den Funktionen verführt werden und eine Bleiwüste präsentieren (so wie hier am Ende meiner Veranstaltung). DAS ist aber nicht eine Frage des Lehrmittels, diesen Fehler kann man immer machen.
Das Notebook ist also für LehrerInnen bzw. DozentInnen ein verführerisches Werkzeug, erlaubt es doch in sauberer, strukturierter Form komplexe Sachverhalte mit verschiedenen didaktischen Kniffs zusammenzufassen und Aufgaben bearbeiten zu lassen. Herz, was willst Du mehr?
So ist es wenig verwunderlich das auch Informatikfachbereiche, die gestern noch Java als Eingangsprache unterrichteten, auf Python umsteigen oder umgestiegen sind und dabei auch auf das Jupyter Notebook setzten. In wieweit sich der Trend fortsetzt oder ob er gar schon wieder rückläufig ist, kann ich an dieser Stelle nicht sagen. Wir können aber aktuell tausende Webseiten rund um das Thema “Jupyter Notebook in the Classroom” finden.
Nach der Lehre
Vor knapp drei Jahren schrieb Jeffrey Perkel in Nature ein Kommentar mit dem Titel “Why Jupyter is data scientists’ computational notebook of choice“. Er beeilt sich die ganzen Vorteile aufzuzählen, die ich auch sehe: Vor allem kann man als DatenanalystIn nicht bloß Ergebnisse in ein Laborbuch kleben, sondern Code, Daten und erklärenden Text in einem Dokument zusammenfassen. Man erhält so ein “computational narrative“, kann aber auch interaktive Tutorials für eigene Software weitergeben. Vor allem lassen sich auf diese Weise auch leichter Veröffentlichungen vorbereiten. Und nicht zuletzt sind die vielen Sprachen, die man nutzen kann und die graphischen Ausgaben und die Möglichkeiten der Anpassung an den eigenen Bedarf sehr nützlich und überzeugend.
Einen Vorteil, den Perkel sieht führt in meinen Augen zu einem riesengroßen Problem: Da es auch möglich ist diese Notebooks mittels “Binder” in der “Cloud” zu nutzen kann man so gleich seine Software veröffentlichen.
Gefahren der Notebooks für die Reproduzierbarkeit
Das Software in der Cloud als Service für Jederfrau/-mann publiziert wird, halte ich für besonders problematisch – und wie der Link zeigt, kommt das inzwischen durchaus vor. Und wissenschaftliche Daten, auch die nicht personenbezogen, haben in “der Cloud” einfach nichts verloren. Sie sind dort nicht sicher vor Verlust. Bei personenbezogenen Daten – auch wenn pseudonomisiert – ist das besonders kritisch, auch wenn einige Staaten das Abladen von personenbezogenen Daten (z. B. menschliche Gensequenzen) nicht verbieten (in Deutschland ist wenigstens dies untersagt und wenig sinnvoll ist es immer). Nicht verschwiegen sei, dass dies viele Menschen anders sehen und die Anleitungen und Hinweise immer mehr häufen (z. B. dieser Artikel mit dem Untertitel “Don’t Just Read it! Do it!“ … nun denn, des Menschen Wille ist sein Himmelreich).
Notebooks verleiten auch zu schlechten Codegewohnheiten, so ist meine Wahrnehmung und Joel Grus vom Allen Institute for Artificial Intelligence, der im Nature-Kommentar zitiert wird, sieht dies ebenso. Grund ist, dass man nicht wie üblich Code logisch ordnet, sondern “einfach” irgendwie weitermacht. Vor allem wird Code nicht in wieder zu verwendende Module gespeichert – oft werden Notebooks wiederverwendet, statt Code sauber zu Wiederverwendung zu versionieren. Konsequenz ist dann auch, dass die Dokumentation vom Vorprojekt nicht mehr passt und sich leichter Fehler bei der Dokumentation einschleichen. Wer notiert denn immer(!) fehlerfrei, kleine Änderungen in den Experimenten, wenn im kopierten Notebook nur wenige Details des Experimentes verändert wurden?
Außerdem sollte ab und an der Kernel des Notebook (also der Interpreter da drin) neu gestartet und der gesamte Code, der in sogenannten Zellen organisiert ist) von oben nach unten ausgeführt werden – nur diese manuelle Überprüfung garantiert, dass sämtlicher Code nach allen Änderungen auch noch funktional ist. Zwar gibt es obendrein Module, welche sämtlichen Änderungen aufzeichnen. In jedem Fall aber ist manuelle Sorgfalt gefragt. Automatisch per default dokumentiert wird da nichts. Reproduzierbare Ausführung und Dokumentation sind so Illusion.
Zu guter Letzt ist interaktive Ausführung in der Datenanalyse immer auch langsam. Während das “Auskundschaften” von Daten und Finden der optimalen Analyseparameter im interaktiven Programmen (nicht nur Notebooks) durchaus angebracht sein kann, sind vielschrittige Analysen mit lang laufen Teilschritten unbedingt zu automatisieren. Sonst passiert ist, dass man in die Mittagspause geht, um zurückzukommen und dann “weiter” zu drücken und erneut zu warten. Produktiv ist anders.
Fazit
Der Einsatz von Jupyter-Notebooks verletzt so ziemlich alle Prinzipien des Software Engineering. Das ist nicht weiter tragisch, doch es entstehen deshalb erhebliche Risiken bei Reproduzierbarkeit der Resultate:
- Es ist nahezu unmöglich gut zu versionieren: Je in einem Team am selben Notebook gearbeitet? Weil die Notebooks nichts anderes als riesige JSON-Strings sind, wenn sie abgespeichert werden, wird das Mergen (=Zusammenfassen via Softwaremanagementsystem) ein Ding der Quasiunmöglichkeit. Darüber hinaus kann man zwar einzelnen Notebooks oder Bündeln davon eine Version verpassen, sie sind jedoch zum Editieren geschaffen: Jeder neue Input, braucht mindestens eine Veränderung.
- Man kann Code beliebig schlechter Qualität schreiben – Jupyter wird sich nicht beschweren. Hauptsache syntaktisch korrekt. Es gibt kein Linting, keine Kontrolle, kein Nichts. Zusammen mit dem nächsten Punkt wird das relevant.
- Jupyter notebooks sind extrem schwer zu testen. Es ist schwer den Code vernünftig zu strukturieren. Und test driven development (TDD) wird unmöglich. Okay, die TDD-Pille ist kein Allheilmittel! Aber eine Umgebung, die TDD erschwert wird auf lange Sicht große kolloborative Projekte verunmöglichen: Wie sonst soll man verschiedene Entwicklungsstränge zusammenführen? Ach ja, die kann es bei Notebooks ohnehin nicht geben, sie sind nicht modularisierbar.
- Nicht-lineare Ausführung kann zu nicht reproduzierbaren Experimenten führen. Da es möglich und oft notwendig ist, zwischen den Zellen des Notebook zu springen, z. B. um einen Irrtum zu korrigieren oder einen neuen Zustand herbeizuführen, hängt der korrekte Zustand eines Notebooks somit manchmal vom fehlerfreien Vorgehen des Anwenders ab.
- Meine Beobachtung ist: Wissenschaftliche Arbeitsgruppen, die Jupyter-Notebooks nutzen, haben mitunter hunderte davon, die für alle möglichen Zwecke, teils redundant, erstellt werden. Sie wandern von Druidencomputer zu Druidencomputer – via EMail, USB-Stick, Gruppenlaufwerk – als Ganzes oder in Teilen. Zusammen mit den Punkten oben ergibt sich: Es ist vielleicht möglich, den Problemen zu begegnen – wird aber nicht einmal versucht.
- Bei langen, asynchronen Aufgaben wird es wirklich problematisch. Big Data bedeutet große Aufgaben. Man fängt häufig an, die Aufgabe in kleine Teile zu spalten, die gut in den Speicher passen. Irgendwann kommt der Punkt, wo das nicht mehr geht. Wer da mit einem Notebook angefangen ist, kann auf Werkzeuge wie Spark zurückfallen. Ich rate dringend davon ab: Besser ist es gleich mit dem parallelen Ansatz anzufangen. Ausbessern, wenn es nicht mehr funktioniert, ist mehr Aufwand und läuft immer mit einer Krücke herum, die fordert: “Jetzt, die nächste Zelle ausführen.”
Da Lernende dazu tendieren dem Beispiele der Lehrenden zu folgen und weil das Notebook eine Sackgasse darstellt, werde ich versuchen davon los zukommen – und es nicht mehr in der Lehre zu verwenden.




Kommentare (16)