Container sind in der Welt der IT keine neue Mode. Und so ist es vielleicht nicht verwunderlich, dass ich vor ein paar Wochen einen atemlosen Anruf eines freundlichen Professors erhielt, der mir mitteilte, wir (also meine Institution) bräuchten dringend eine Cloud mit einem bestimmten containerbasierten Workflowsystem. Anders sei reproduzierbare Bioinformatik heutzutage nicht mehr darstellbar! Und auch an HPC-Konferenzen geht das Thema Cloudcomputing nicht vorbei. Also, was sind eigentlich Container in der IT? Wo sind sie nützlich? Was bieten sie – und was nicht? Und führt am Thema Cloudcomputing im wissenschaftlichen Rechnen wirklich kein Weg vorbei? Schauen wir uns die Sache einmal an:
Was sind Container? Wozu braucht man sie?
Allüberall werden Container eingesetzt und vielen Admins hängt das Containerlied der Hersteller und Wissenschaftler – die unbedingt Container brauchen!!! – schon zum Hals raus. Denn wer hier nicht einstimmt, gilt als Innovationsverweigerer. Es ist also ein aufgeladenes Thema, wie so oft in der IT.
Container sind Virtualisieurngslösungen – das heißt, man kann alle mögliche Software, inklusive ganzer Betriebsysteme und ihre Paketbasis, in einen Container packen und den Container dann so ausführen als seien die darin verpackten Programme in einer anderen Umgebung als in dem System, von dem man sie gerade ausführt. Das große Versprechen ist denn auch der Abhängigkeitshölle, also dem Umstand, dass man für eine lauffähige Software tonnenweise andere Software installieren muss (in den jeweils richtigen Versionen; zur Bedeutung siehe auch hier), entkommen kann.
Container – bzw. “containerisierte” Anwendungen – werden im wissenschaftlichen Rechnen vor allem in Cloud-Umgebungen eingesetzt. In einer ziemlich spartanischen eingerichteten virtuellen Maschine einer Cloud kann man so einfach seine Software mitnehmen und ist sich sicher: Das wird laufen. Und Cloud-Computing ist in den Naturwissenschaften “in”. Mit steigender Tendenz werden die Begriffe “Cloud” und “bioinformatics” in der Metadatenbank wissenschaftlicher Veröffentlichungen, PubMed, denn auch mit Publikationen verknüpft:
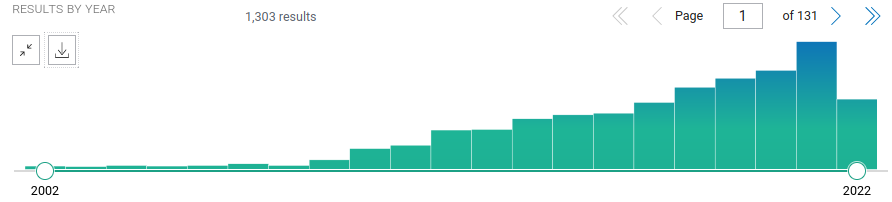
Screenshot einer Suche mit den genannten Begriffen vom 14. August 2022.
Die Schlussfolgerung, dass sowohl Cloud-Computing als auch Container im lebenswissenschaftlichen Rechnen ebenso immer bedeutsamer werden, ist also zulässig. Insbesondere im angelsächsischen Raum, gibt es die Tendenz keine eigene Rechenleistung mehr vorzuhalten, sondern das Geld lieber Google, Amazon, Microsoft und anderen Cloud-Betreibern zu geben. Der Grund hierfür ist simple: Man glaubt so Geld sparen zu können. Weniger Personalkosten, Wartungskosten, Unterhaltskosten sind sicher ein guter Grund. Hierzulande setzt man eher auf föderale Lösungen.
Bezahlt wird nicht nur mit Geld
Wenn man nur mit Geld bezahlt werden müsste … Nein, auch mit Vertrauen muss man zahlen: Sind meine Daten in der Cloud sicher? Das ist doch die alles bestimmende Frage. Und ich bin froh, dass hierzulande das Auslagern in x-beliebige Clouds aus Datenschutzgründen nicht zulässig ist, vor allem bei humangenetischen Daten ist es nicht geraten bzw. verboten, außerhalb der EU derartige Daten zu speichern. Geldmittelgeber sehen auch die Anlage von Drittmitteln in Anbetracht existierender föderaler Infrastruktur für wissenschaftliches Rechnen nicht gerne. Misstrauen ist ohnehin angebracht.
Es geht hier aber nicht im Kern um Cloud-Computing. Das ist nur ein häufiger Anwendungsfall für Container. Container sind auch sonst praktisch, denn sie bringen ihr eigenes Userland mit (man kann also recht vertraut mit ihnen arbeiten) und in puncto Sicherheit gibt es auch eine Hoffnung, die sich mit ihnen verbindet: läuft ein Container auf einem (potentiell verwundbaren) (Web-)Server und eine Attake ist erfolgreich, so bleibt der Angreifer in dem Container gefangen und kann nicht heraus. Diese Sicherheitsebene fehlt ohne Container.
Letztlich jedoch kommt es bei Containern auch darauf an, wer Urheber ist und ob dieser vertrauenswürdig ist. Installationsframeworks (easybuild, spack etc.) und Paketverwaltungen der großen Distributoren (Ubuntu, Debian, SuSe, etc.) haben dieses Problem gelöst durch verschiedene Maßnahmen (signierte Pakete, Checksummen für jede Abhängigkeit). Die Situation bei Containern ist ein wenig anders. Die großen Hersteller bieten zwar signierte Basis-Images an, doch diesen fehlen bestimmte Dienste. Also müssen Anwender und Administratoren oft andere Wege gehen.
Eine Möglichkeit ist ein Continous Integration und Deployment-System (CI/CD) wie z. B. Jenkins zu nehmen. Die Idee: bei Erscheinen eines neuen Softwarerelease wird automatisch ein neues Image generiert. Das “Ausrollen” müssen Admins nur noch per Mausklick bestätigen. Damit jedoch bezahlt man entweder für ein fertiges, professionelles System oder – im akademischen Fall weitaus häufiger – man bastelt selber und bezahlt mit der Zeit, die man besser mit Forschung verbringen könnte. CI/CD-Systeme sind ohnehin keine Selbstläufer und produzieren einen gewissen Aufwand. Im wissenschaftlichen Rechnen werden Container folglich oft handgeklöppelt. Der Umstand, dass es Containersysteme gibt, die es ebenfalls erlauben signiert zu werden, hilft nicht weiter, denn wo der Laie Container bastelt, wird darauf sehr häufig verzichtet.
Und auch die Sache mit dem Entkommen aus Abhängigkeitshölle stellt sich bei vielen Containern auch nicht so einfach dar, wie man zunächst denken könnte. Wer im Container nur auf Pakete eines Providers angewiesen ist, ist fein raus. Wissenschaftliche Anwendungen bringen jedoch häufig versionsspezifische Abhängigkeiten zu anderen wissenschaftlichen Anwendungen oder Bibliotheken mit – viele! Da wird das Bauen von Hand nicht viel einfacher, als es unabhängig vom Container mittels Installationsframework zu versuchen.
Und das Handklöppeln von Containern bedeutet auch, dass im wissenschaftlichen Zusammenhang verwendete Software oftmals nur über Trampelpfade und inoffizielle Quellen zu erhalten ist. Dass die Reproduzierbarkeitskrise in den Neurowissenschaften (und Psychologie sowie Psychatrie) besonders ausgeprägt ist, ist bekannt. Die Gründe auch (Studiendesign, Jagd auf bessere p-Werte, etc. etc. vieles wurde schon lange auf Scienceblogs diskutiert). Was hinzu kommt ist, nachdem ich jahrelange Erfahrung mit dem Feld auf der Softwareseite habe, ist der Umgang mit Software und Daten (teils hier im Blog bereits beschrieben). Ihr kennt bestimmt alle die Geschichte vom toten Lachs, dessen fMRI-Daten analysiert wurden und schön interpretierbare Daten lieferten – wegen multiplen Testens auf verrauschen Daten. Nun, die Interpretation von Daten ist tägliches wissenschaftliches Brot und die Software, die zur Analyse von MRI-Daten beliebt ist, ist ein Problem an sich. Als, nicht zum ersten Mal, eine ganze Community das Installationsproblem nicht lösen konnte und Kunden warteten, habe ich die Entwickler angeschrieben. Sie konnten nicht weiterhelfen, wollten ohnehin ihr Installationsschema komplett umstellen, weil sie selber nicht mehr durchblickten (die vorgeschlagene Lösung ließ mich in die Tischkante beißen, aber das ist eine andere Geschichte). Und freundlicherweise erhielt ich diese Antwort:
I’m very sorry to hear you’re having problems compiling FSL. As an interim solution, I can provide you with a Singularity image that will correctly install FSL6.0.4
So haben wir es dann gemacht. Schön ist das nicht, aber es funktionierte.
Immer häufiger wird wissenschaftliche Software nur mehr in Containerform angeboten. Da bekommt das “but it runs on my system!”-Argument, mit dem Entwickler den Report von Problemen gerne abwiegeln (weil: “Bei mir funktioniert es!1!!11!!”), eine ganz neue Qualität. Eine wirkliche Lösung ist das jedoch nicht, denn man muss zum Teil manch forensische Energie reinstecken, um herauszubekommen, welche Bibliotheken und welche Versionen denn im Container stecken. Klar, wenn ein Container gegeben ist, kann man den Container mit den Daten archivieren und ist sich “sicher”, dass die Daten damit für alle Zeiten reproduzierbar bleiben (vorausgesetzt, der Container ist auch in ferner Zukunft lauffähig und wir lassen all die lästigen Argumente über sich verändernde CPU-Befehlssätze außer acht), oder? Na, wer sagt einem, dass Version X einer Software im ersten Container dieselbe Software ist, wie die Software X derselben Software in einem anderen Container? (Wenn die Checksummen unterschiedlich sind und die Dinger nicht signiert sind, kann das tausendundeine Ursache haben.) Und dass die Versionierung überhaupt stimmig ist? Wie es um derartige Versprechen bestellt ist, muss ich nicht mehr beschreiben. Fleißige Mitleser wissen Bescheid, oder?
Und noch eine Überlegung zu der Frage, womit bezahlt wird. Schauen wir uns kurz folgenden Vergleich an:
- bei einer nativ installierten Anwendung erhalte ich eine Antwort auf die Hilfeanfrage (also so was wie
$ time cmd --help) in ungefähr 1-4 Sekunden (hängt ab von Caches, Reponsivität des Filesystems, größe der Anwendung und was diese sonst noch so macht, bis sie eine Antwort ausspuckt, ab) - bei einer in einem Singularity-Container eingepackten Anwendung erhalte ich dieselbe Antwort in 70-80 Sekunden (weil der Container erst in eine sog. “Sandbox” entpackt wird)
Eine derartige Containeranwendung zu starten, bringt also einen gewissen Overhead mit sich. Bei parallelen Anwendungen fällt dieser Overhead mit der Zahl der reservierten Kerne ins Gewicht – Teile des Computers, die däumchendrehend Strom verbrauchen. Also eine Zahl irgendwo zwischen 2 und ein paar Tausend. Da die immer gleichen Anwendungen viele Tausend mal eingesetzt werden und Container für tausende Anwendungen existieren, kommt man so leicht auf Millionen verschwendeter CPU-Stunden. Keine Kleinigkeit in diesen Zeiten, auch wenn die Performance der Anwendung nach dem Start nicht schlechter ist als die einer nativen Anwendung (was aber keineswegs garantiert ist).
Wie steht es nun um das letzte Versprechen der Container-Jünger? Das, das da lautet: “Wir packen die Anwendung (ggf. sogar mit Daten) in einen Container, dann können wir unsere Ergebnisse bis in alle Zeiten reproduzieren!” Solcherlei Aussagen (und man hört sie gelegentlich, wenn auch immer wieder anders formuliert) lassen mich manchmal sprachlos zurück. Richtig ist, dass containerisierte Anwendungen leichter portierbar sind und wenn die verwendeten Algorithmen deterministisch sind (ihr Ergebnis also beispielsweise nicht von Zufallskomponenten abhängt) ist das Einpacken in Container eine gute Möglichkeit zu archivieren und zu portieren, kurz: durchaus ein Schritt zu größerer Reproduzierbarkeit.
Leider weiß so ein Container auch nicht, welches die neuesten Moden der CPU-Herstellen in 10 Jahren sein werden. Und auch prinzipiell deterministische Algorithmen leiden manchmal unter unterschiedlicher Genauigkeit der Prozessoren und dann macht die mathematische Fehlerfortpflanzung einen Strich durch die Rechnung (sorry: pun intended). Und auch von solchen Details abgesehen: ohne ausreichende Metadaten (z. B. welche Parameter wurden warum für welche Daten gewählt) ist es mit der Reproduzierbarkeit einfach nicht weit bestellt. Auch die Anwendbarkeit leidet manchmal, wenn im Container Voreinstellungen hart verdrahtet und gegenüber den Anwendern nicht transparent dokumentiert sind.
Fazit
Container sind eine großartige Technologie, auch für wissenschaftliche Anwendungen überaus nützlich und manchmal können wir in der Forschung nicht darauf verzichten. Das hohe Lied auf die Container klingt dennoch manchmal schief: die Fürsprecher der stärkeren Containerisierung für wissenschaftliches Rechnen im Allgemeinen sollten sich gelegentlich mal anschauen, ob ihre Argumente überall und uneingeschränkt gelten. Und wenn nicht: tief durchatmen, es gibt hier und da auch andere Möglichkeiten. Doch wenn es ein Container sein soll und die Anwendung liefert solide Ergebnisse? So sei es! Geht auch auch ohne Rumreiten auf Sicherheit und Reproduzierbarkeit und die Behauptung, dass Container allein selig machend sind.



Kommentare (7)