Vor ein paar Tagen war ich schlaflos, griff ins Regal und fand das schöne Buch “Die Logik des Misslingens” (kommerzieller Link) des Psychologen Dietrich Dörner. Ein schönes Buch, auch wenn schon etwas älter. Und bei einem Kapitel fiel es mir wie Schuppen von den Augen …
Warum haben Aliens menschliches Antlitz?
Dörner wundert sich, warum beim Gefecht bei Langarde im Ersten Weltkrieg das Ulanen-Regiment noch eine Kavallerieattacke ritt. Konnte man sich nicht vorstellen, dass es keine gute Idee ist, mit Pferden Maschinengewehrstellungen anzugreifen? Nein, argumentiert er, man konnte nicht. Bei Beginn des Krieges zog man noch trommelnd und pfeifend, die “Offiziere mit gezogenem Degen”, zur Front. Der Krieg, den man erwartete, war eine Neuauflage des deutsch-französichen Krieges von 1870/71. Den Stellungskrieg, der folgen sollte, konnten sich die meisten noch nicht vorstellen.
Für mangelnde Vorstellungskraft gibt es im Buch noch weitere Beispiele: Erste Autos, die Pferdewagen ohne Pferden glichen. Oder Jule Vernes Fantasiereise zum Mond, gestartet mit einem ballistischen Projektil (obwohl Raketen als Feuerwerkskörper längst bekannt waren). Und die Aliens in der Science Fiction, die häufig menschenähnlich (vier Extremitäten, meistens zwei Augen, Nase, etc.) erscheinen. All dies, weil die Vorstellungskraft nicht reichte, über bekannte Strukturen hinaus zu denken.
Und ich merke, trotz vieler Beispiele im Blog, manche regelmäßige Kommentatoren können sich oft gar nicht vorstellen, was mit “wissenschaftlicher Software” so gemeint sein könnte. Und die Ursache für all dies? “Strukturextrapolation!”, so meint Dörner für seine Beispiele erkannt zu haben. “Strukturextrapolation!” vermute ich als Ursache für das Verständnisproblem, das manche LeserInnen des Blogs haben.
Bei der Strukturextrapolation stellt man sich zukünftige oder bislang unbekannte Sachverhalte so vor, wie entsprechende, bereits bekannte. Deshalb haben manche Ingenieure Schwierigkeiten zu akzeptieren, dass ihre Herangehensweise ungeeignet ist, die “Weltformel” zu finden. Deshalb wissen Eltern, wie Schule zu funktionieren hat (und stoßen manchmal LehrerInnen vor den Kopf). Deshalb erklärt einem der Arzt aus der Nachbarschaft die Epidemiologie, denn er kennt ja die Zahlen aus seiner Praxis (und weiß seit Jahren, dass die Krankenkassen Unsinn publizieren).
Die Liste der Beispiele ließe sich endlos fortsetzen, wobei man vorsichtig sein muss, weil Strukturextrapolation keine umfassende Erklärung für alle möglichen Verständnisprobleme ist – und außerdem können wir uns alle in diesem Fallstrick verheddern. Dennoch glaube ich mich genau diesem Problem regelmäßig gegenüberzusehen, wenn ich meine Einführungsveranstaltung für die Nutzung von Hochleistungsrechnern mache: Ich weiß genau, dass meine Klientel allenfalls Berührung mit Laptops, Desktop-PCs und im besten Fall 0815-Servern hatte und entsprechen formulieren die Leute anfänglich ihre Fragen. Im Lauf von zwei Tagen schaffe ich es meist, den Unterschied zum und Sinnhaftigkeit des Rechnens auf Supercomputern zu vermitteln.
Was also ist “wissenschaftliche Software”?
An dieser Stelle ist es natürlich besser kleine Brötchen zu backen: Zu versuchen alle Verständnisprobleme, die durch Strukturextrapolation geschaffen wurden aus der Welt zu räumen ist ein Ding der Unmöglichkeit – es gibt ja hier im Blog und anderswo noch mehr Gelegenheiten.
Worum geht es also, wenn hier von “wissenschaftlicher Software” die Rede ist? Zunächst einmal geht es, etwas präziser, um Software, die verwendet wird um wissenschaftliche Fragestellungen zu beantworten. Und das kann von der Natur her eigentlich allem gleichen, was man selber schon irgendwo kennengelernt hat. Hier im Blog hatten wir schon Webtools in der Bioinformatik und auch grafische Benutzeroberflächen kamen schon kurz einmal vor. Hier zu kategorisieren und zu zählen, was am häufigsten vorkommt, ist nicht einfach.
In der biomedizinischen Mikroskopie dominieren grafische Benutzeroberflächen (Beispiel), viele kommerziell. Gerne verbinden Firmen auch für andere Zwecke Messapparate mit dem Verkauf von Steuer-PCs, um ein Komplettangebot (Steuerapparatur, Messapparat und Auswertesoftware) anbieten zu können. Durchaus sinnvoll und meist, denn die Nutzerbasis erwartet das, unter Windows (Beispiel, Beispiel, Beispiel). Das geht bis zu ganzen Labor-Informations- und Management-Systemen (Beispiel). Die Liste der Beispiele könnte hier noch lange weitergehen und doch wäre es mir nicht möglich eine vollständige Liste zusammenzufassen: Der Versuch das zu tun und zu verallgemeinern wäre Strukturextrapolation meinerseits, denn ebenso wie meine Leserschaft habe ich einen beruflichen Fokus beziehungsweise eine Berufsdeformation und kenne gar nicht die gesamte Welt der wissenschaftlich-technischen Informatik*. Dennoch können wir an dieser Stelle zu einer allgemeinen Aussage gelangen, nämlich, dass die meiste Software auf Linux läuft, bzw. auf linuxoiden System die meisten CPU-Stunden verbraten werden / die meiste Analysezeit erfolgt.
Warum? Weil es “einfach” ist, unter Linux Software zu entwickeln. Vor allem kostet es nicht viel, da wenigstens hier freie, quelloffene Software, inklusive gängiger Entwicklungswerkzeuge dominieren. Vor allem aber laufen alle Feld-, Wald- und Wiesenserver zur Datenanalyse und auch viele, viele Cloudinstanzen zum selben Zweck unter Linux. Nicht zuletzt sind hier sämtliche Supercomputer der Welt zu zählen (vgl. Top500-Liste), wo so unglaublich viele CPU-Stunden zusammenkommen, dass die Zahl der Nullen einem Astronomieblog Konkurrenz machen kann.
Hier geht sehr viel Zeit auf Simluationprogramme und Datenanalysen. Simulationsprogramme sind “einfach”: Simulation durchführen, auswerten, fertig. “Einfach” steht hier natürlich in Anführungsstrichen, weil das Programm an sich und die Anwendung mitunter auch selbstverständlich nicht einfach sind, aber die Abfolge der Schritte ist wenig komplex. Wie sieht das aus?
gmx mdrun -deffnm $configfile
Nicht mehr als eine Befehlszeile in einem Script. (Der Programmname ist mit einem Link hinterlegt, alle Dinge mit ‘$’ stehen für Variablen, hier also für die Angabe einer Datei, in der die Konfiguration für eine bestimmte Molekulardynamiksimulation steht.) Visualisierung der Ergebnisse und damit ein interaktiver Teil ist wichtig! Darum geht es schließlich. Solcherlei Programme, in vielen, vielen Ausprägungen, dominieren die insgesamt benötigte Rechenzeit. Die Visualisierung findet später statt und braucht vergleichsweise wenig Rechenpower.
Datenanalysen mit Präprozessierung (z. B. Qualitätschecks, Artefaktentfernung, ggf. Imputation, eigentliche Rechnung(en), Zusammenfassung) kann beliebig komplex werden. Als Beispiel soll ein kleiner(!) Workflow dienen, der eine Auswertung für Transkriptom-Sequenzierung ergibt – aber im Grunde genommen kann so was auch in der Pharmazie oder Teilgebieten der Experimentalphysik ganz ähnlich vorkommen.
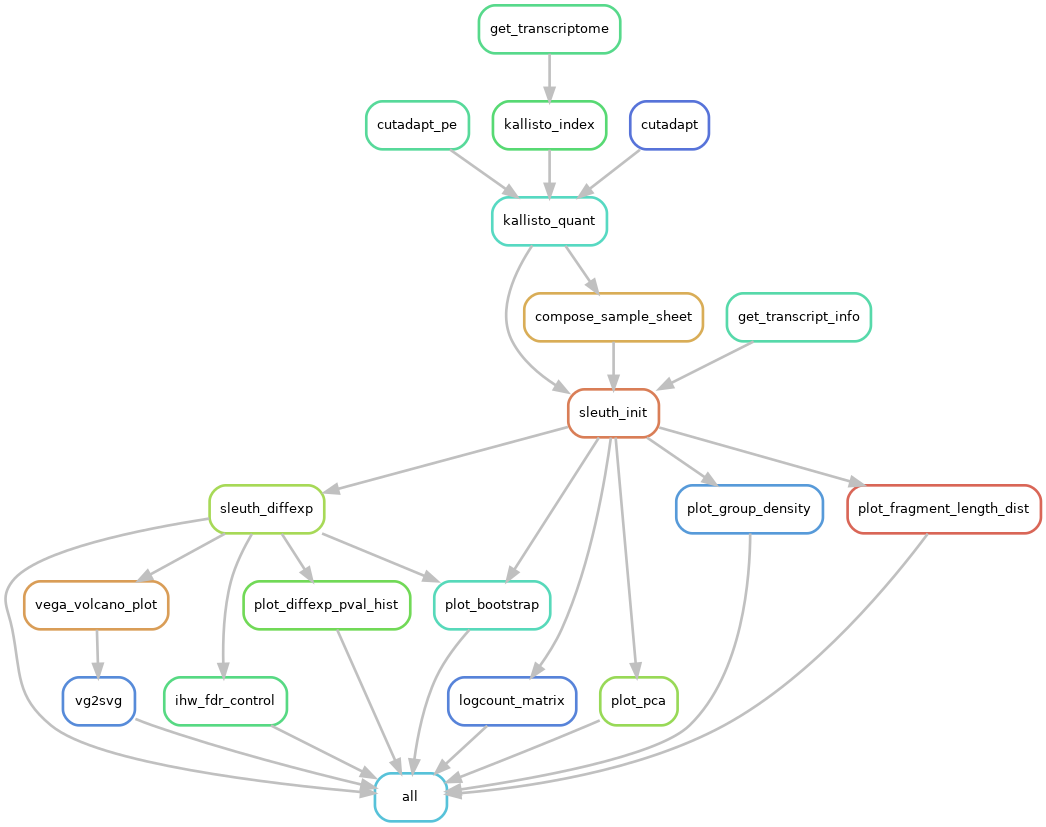
Jeder einzelne Kasten steht für einen Analyseschritt – die Bezeichnungen erschließen sich eher den Anwendern. Wesentlich für den Workflow ist eine Eigenschaft, die durch die Abbildung nicht deutlich wird: Er ist skalierbar. Das heißt, bestimmte Analyseschritte werden bei mehr Eingabedateien mehrfach ausgeführt. (Das ist für unseren Punkt nicht wichtig. Aber für mitlesende BioinformatikInnen, die daran denken “Pipelines” zu publizieren, möchte ich das nicht unerwähnt lassen.) Uns interessiert die verwendete Software, also schauen wir uns mal das zentrale Programm im “kallisto_quant”-Schritt an. Es wird z. B. so aufgerufen (im Detail können und werden Programmaufrufe abweichen):
kallisto quant -i $input.idx -o $output
Auch hier: Ein Aufruf in einem Script mit einem Eingabe “$input.idx” und einer Ausgabe “$outupt”. (Und noch eine für AnwenderInnen nicht unwichtige Eigenschaft des Workflows ist: Er abstrahiert alle Programmaufrufe und kümmert sich um Setzen von Parameter, Ein- und Ausgaben).
Visualisierung ist wichtig!
Alle diese Software läuft ohne, dass man zuschauen kann, was passiert – die Ausgaben geschehen in (z. T. nicht menschenlesbare) Ausgabendateien. Abgesehen von Randgebieten spielt real-time-computing, also Software zur unmittelbaren Aufnahme oder Steuerung von Prozessen, keine Rolle. Ein Beispiel für ein solches Randgebiet ist die eingangs genannte Mikroskopie oder auch Messrechner in der Experimentalphysik. Ich zum Beispiel habe die Kleinwinkelstreudaten (boah, ist der Wikipediaeintrag dünn!), Neutronenstreudaten und optische Daten für meine Dissertation auch “live” aufgenommen (lies: den Detektoren, beim Detektieren mittels graphischem Display zuschauen können). Die dafür notwendigen Applikationen sind auch alles relevante Programme im Sinn “wissenschaftlicher Software” – doch Datenauswertung und Simulation geschah in allen mir bekannten Bereichen zumeist nicht interaktiv. Wissenschaftliche Simulation und Datenanalyse ist zum allergrößten Teil Stapelverarbeitung – in all ihren Ausprägungen (der verlinkte Wiki-Artikel liest sich etwas altbacken, aber korrekt) – es geht also um automatisierte Übergabe von Ein- und Ausgabedateien um Zeit zu sparen.
Okay, bei Finite-Elemente-Simulationen schaut man sich gelegentlich durchaus an, was gerade simuliert wird. Und vielleicht denkt ihr an eure Arbeit und es zuckt schon in den Fingern: “Aber ich weiß, dass es anders ist, viel visueller und interaktiver!” Kein Zweifel, das wird hier nicht in Abrede gestellt! In meinem Beispiel und häufig bei ähnlichen Anliegen werden Plots durch Skripte bei Auswertung der Workflowergebnisse erstellt. Und, wie gesagt, der Artikel erhebt keinen Anspruch auf eine voll umfassende Beschreibung, sondern auf eine Beschreibung der wesentlichen und häufigsten Anwendungstypen von Software in der Wissenschaft.
Unterschlagene Moden
Was jetzt völlig unterschlagen wurde, sind die großen Moden der “computational science”:
- künstliche Intelligenz – die nenneswerten Anwendungen sind ebenfalls alle zwar mit Visualierungstools versehen, aber i.d.R. auf der Kommandozeile zu instrumentarisieren und die Hauptrechenzeit findet “unsichtbar” statt (Beispiele: Tensorflow/pyTorch, sklearn)
- Parallelisierungstools für anderweitig nicht oder nicht hinreichend parallelisierte Anwendungen (für “loosly coupled problems” für die Fachleute) – Hadoop/Spark (nennenswert, wenn auch anderer Natur: GNU parallel)
Dies sind auch alles Techniken, die auf Anwenderseite zumindest Skriptingfähigkeiten voraussetzen. Auch da gibt es, im Blog beschriebene, Ausnahmen, die jedoch allenfalls Nischen besetzen.
Und wie findet man solche Software?
Die Informatik publiziert zuweilen in Konferenzzeitschriften und Tagungsbänden. Andere Wissenschaften überwiegend in Fachzeitschriften (wie es darum in der Bioinformatik bestellt ist und warum das ein Problem ist, kann man gerne nochmal nachschlagen). Dann, mit ein wenig Glück, liegt der Quellcode auf einer Hostingplattform einer Uni oder auf github. Mit ein wenig mehr Glück kann man dann auch herunterladen und nutzen. Geht halt nicht immer. Doch obwohl solch ein Schrott – Verzeihung! – an der Publikationstagesordnung ist, bestimmt er zwar das Publikationsgeschehen, aber nicht das Bild der Anwender – aus guten Gründen. Was funktioniert mag Fehler enthalten, aber nur was wenigstens halbwegs funktioniert, findet Anwendung in der Breite.
Zum Abschluß
Wer jetzt eine Definition erwartet hatte, wurde enttäuscht. Es hilft nur den vielen Links nachzugehen, in diesem Artikel und den verlinkten Artikeln zum Blog und natürlich auf github** – oder sich wirklich tief, tief einarbeiten in bestimmte Felder. Software in den Naturwissenschaften dominiert in Zahl der Veröffentlichungen alle anderen Wissenschaften und mag von Feld zu Feld sehr unterschiedlich sein. Der gemeinsame Nenner der Communitysoftware ist häufig: Kommadozeilenorientierung, läuft nicht auf Windows (es sei denn unter WSL oder eben in irgendwelchen portablen Skriptsprachen geschrieben) und man muss Dokumentation lesen, um sie anzuwenden – Nutzerfreundlichkeit geht häufig anders.
Man darf sich also klar machen: Alles was auf Anwenderseite ein Übermaß Gefrickel voraussetzt findet keine Verbreitung und ist somit wenig repräsentativ. So kenne ich von früher™ Leute, die Fouriertransformationen en masse mittels Postscript auf Druckerkarten rechneten – war billig, aber nicht portierbar; nie wieder was davon gehört. Wie wir unlängst lernten kann man auch via Javascript auf Excel(dateien?) zugreifen und rechnen – auch das wird wohl eher keine weite Verbreitung finden. Was irgendwo(!) irgendwie(!) funktioniert ist nicht repräsentativ für irgendeine Art von Software, inklusive aller anderen genannten und ungenannten Ausnahmen. Was keine weite Verbreitung findet (mindestens tausende Installationen) ist eine Ausnahme. Vorsicht vor Strukturextrapolation!
++++
- Insbesondere ist mir bewusst, dass Ingenieursbüros oftmals andere Software einsetzen, eine andere Herangehensweise an Datenanlysen pflegen und wenig von einer Welt (wissenschaftliche Datenanalyse) in die andere (technische Anwendungen) übertragbar ist. Strukturextrapolationsfallstricke lauern auch umgekehrt.
** Ja, auch diese Plattform darf man, zu recht, kritisch sehen.



Kommentare (9)